В комментариях к первой части статьи один из читателей нашел яркие слова и написал – «такое не делает больше никто в мире». Не будем играть в ложную скромность – читатель прав, никто пока подобного не делал. Есть всего один человек, который может сделать, и он готовит одну (как минимум) из следующих статей на Переформате по персональным интерпретациям – это Игорь Львович Рожанский. Если у него, по мнению читателей, получится лучше – я буду только рад.

В этой части речь пойдет о гаплогруппе I. Это – вторая по численности (после R1a) гаплогруппа среди этнических русских, гаплогруппа с трагической древней историей, которая (и гаплогруппа, и история) ускользнула от внимания историков и археологов. Только ДНК-генеалогия и ископаемые ДНК позволили приоткрыть эту завесу, но с разных сторон. Ископаемые ДНК находят с датировками 7-5 тысяч лет назад, а ДНК-генеалогия показала, что подавляющее большинство современных носителей гаплогруппы I1 по всей Европе имеют общего предка, который жил всего 3700 лет назад, а современных носителей гаплогруппы I2a в Восточной Европе – только 2300 лет назад. Описываемые здесь и в последующих частях статей по персональным интерпретациям примеры углубляют и расширяют это новое знание. Еще в этой части мы начинаем описание персональных интерпретаций мтДНК. Об этом подробнее – ниже. По всем вопросам касательно персональных интерпретаций пишите на электронный адрес Академии ДНК-генеалогии: info@dna-academy.ru
 Все выпуски международного научного журнала «Исторический формат» можно бесплатно скачать на официальном сайте. Сейчас готовится к изданию третий номер. Это свободный и открытый проект в стиле Open Science, который имеет научные и просветительские цели. Примите прямое и деятельное участие в выпуске журнала «Исторический формат», поддержите первое в России независимое научное издание по истории, войдите в состав Общественного попечительского совета журнала! Подробнее… Все выпуски международного научного журнала «Исторический формат» можно бесплатно скачать на официальном сайте. Сейчас готовится к изданию третий номер. Это свободный и открытый проект в стиле Open Science, который имеет научные и просветительские цели. Примите прямое и деятельное участие в выпуске журнала «Исторический формат», поддержите первое в России независимое научное издание по истории, войдите в состав Общественного попечительского совета журнала! Подробнее… |
Эту гаплогруппу имел всего один человек, причем с 12-маркерным гаплотипом.
KLIN ID00020
Был представлен 12-маркерный гаплотип, и сообщено, что он относится к снипу I-М253: 13 22 14 10 14 14 11 14 11 12 11 28. Это – гаплогруппа I1, но показанный снип дает ее самую поверхностную характеристику (см. диаграмму снипов гаплогруппы I1 ниже). На этой диаграмме находится общая гаплогруппа/субклад М253, которой около 30 тысяч лет, и, естественно, от нее отходят десятки нисходящих субкладов. На диаграмме оставлены только те, которые наиболее вероятно относятся к представленному гаплотипу (отмечены желтым цветом), и их ближайшие соседи, как показали исследования в этой Интерпретации. Ниже будет показано, как мы пришли к этим выводам, поскольку сам представленный гаплотип и его снип M253 сами по себе почти никакой информации не дают.
В правой колонке ниже представлены (в годах, лет назад) примерные датировки возникновения древнейших и некоторых других снипов, которые здесь представляют для нас интерес, и рассчитанные компанией YFull по геномным данным.

Как видно, ДНК-линия заказчика наиболее вероятно пошла со времен 3600-4700 лет назад, то есть со времен возникновения снипов, наиболее вероятных у ID00020. Проверим это. Попытаемся определить место представленного гаплотипа среди всех 968 111-маркерных гаплотипов гаплогруппы из Проекта I1 FTDNA.
111-маркерные гаплотипы были взяты как наиболее детальные, и оказалось, что среди них имеются 15 гаплотипов с первыми 12-маркерами точно такими же, как в представленном гаплотипе. Это гаплотипы с номерами 57-60, 110, 131, 132, 153-157, 660, 744, 745 по списку из 968 111-маркерных гаплотипов гаплогруппы I1. Далее было построено дерево из всех 968 111-маркерных гаплотипов, с помощью профессиональной компьютерной программы (PHYLIP), и наиболее родственные (по первым 12 маркерам) гаплотипы были выявлены. Полученное дерево приведено ниже, для иллюстративных целей. Оно показывает хорошую симметричность, что говорит о том, что все дерево происходит от одного общего предка. Когда он жил – будет также показано. Для сведения, базовый (предковый) гаплотип всего дерева следующий
13 22 14 10 13 14 11 14 11 12 11 28 – 15 8 9 8 11 23 16 20 28 12 14 15 16 – 10 10 19 21 14 14 16 20 35 37 12 10 – 11 8 15 15 8 11 10 8 9 9 12 23 25 15 10 12 12 16 8 13 25 20 13 13 11 12 11 11 12 11 – 32 12 8 17 12 24 27 19 11 12 12 13 11 9 11 11 10 12 12 31 11 13 21 16 11 10 24 15 19 11 24 17 13 15 25 12 22 18 12 14 18 9 12 11
Представленный гаплотип в первых 12 маркерах отличается от него всего на одну мутацию (пятая мутация по счету, выделено). Всего же все 968 гаплотипов показывают 14891 мутацию от первой 67-маркерной панели, и 24990 мутации на всех 111 маркерах. Это показывает, что общий предок жил 14891/968/0.12 = 128 → 147 условных поколений (по 25 лет), или 24990/968/0.198 = 130 → 149 условных поколений (0.12 и 0.198 – константы скоростей мутаций для 67- и 111-маркерных гаплотипов, стрелка – табличная поправка на возвратные мутации), или 3675±370 и 3725±375 лет до общего предка, соответственно. Видно, что 67- и 111-маркерные гаплотипы дают практически идентичные результаты, с разницей всего на 50 лет на фоне 3700 лет.

Оказалось, что указанные семь гаплотипов, содержащие такие же 12 маркеров/аллелей, находятся в трех разных ветвях дерева. Конечно, были бы в представленном гаплотипе 25, 37 маркеров или больше, определенно выявилась бы только одна ветвь, но что есть, то есть. В принципе, это нам помешает не сильно. Эти ветви – следующие:

Первая ветвь, в ней один гаплотип из перечисленных, под номером 58.
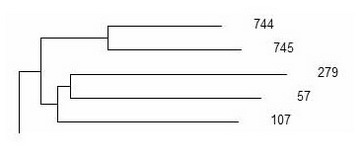
Вторая ветвь, включает гаплотипы 57, 744 и 745.

Третья ветвь, включает гаплотипы 59 и 60.
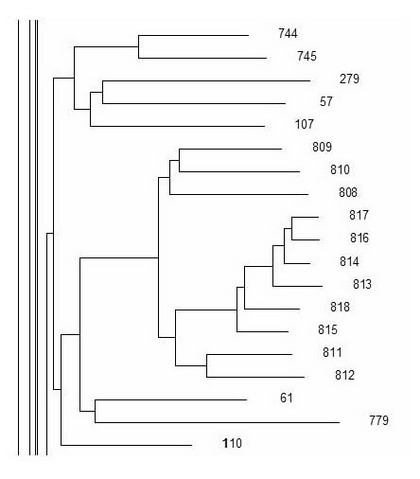
Последняя ветвь (из 19 гаплотипов) – сводная, она названа здесь четвертой, но включает полностью вторую ветвь (с гаплотипами 57, 744 и 745), плюс гаплотип 110. Базовые (предковые) 12-маркерные гаплотипы ветвей, соответственно:
13 22 14 10 13 14 11 14 11 12 11 28 — 1600±240 лет до общего предка
13 22 14 10 14 14 11 14 11 12 11 28 — 2960±410
13 22 14 10 14 14 11 14 11 12 11 28 — 1570±225
13 22 14 10 13 14 11 14 11 13 11 29 — 3150±350
(три мутации от представленного гаплотипа выделены). Представленный гаплотип в принципе может быть в любой из этих ветвей, его всего лишь 12 маркеров не позволяют исключить ни одну из четырех ветвей. Общие предки этих четырех ветвей жили, как указано справа от предковых гаплотипов.
Посмотрим, к каким субкладам относятся родственные представленному гаплотипы под приведенными выше номерами, и у которых полностью повторяются его первые 12 маркеров. В конце данного материала приводятся геномные данные по четырем из этих человек, в которых отмечены снипы, нижестоящие от M253. Скорее всего, эти же снипы будут у ID00020. В целом же снипы у большинства их этих гаплотипов: I1-M253 > DF29 > CTS6364 > M227 и в другой ветви I1-M253 > DF29 > Z58 > Z138. Снипы ID00020 должны относиться или к первой или ко второй цепочке. Все они будут определяться в московской Лаборатории ДНК-генеалогии.
Исторический путь к представленному гаплотипу примерно следующий. Гаплогруппа I образовалась около 43 тысяч лет назад (из сводной гаплогруппы IJ), около 28 тысяч лет она разошлась на гаплогруппы I1 и I2 (хотя «разошлась» здесь понятие условное, они не разошлись, это были независимые события), из первой (как и из второй) с течением времени образовалось множество субкладов. Гаплогруппа I была найдена в ископаемых скелетных останках с датировкой 7 тысяч лет в Швеции и Центральной Европе. В интервале 4600-4000 лет назад, в ходе заселения Европы эрбинами, носителями гаплогруппы R1b, гаплогруппа I1 из Центральной Европы практически полностью исчезла, но кто-то выжил, и как результат этого современные носители гаплогруппы I1 имеют относительно недавнего общего предка, который дал выживших потомков примерно 3700 лет назад, то есть в первой половине II тыс. до н.э. Поскольку общий предок в гаплогруппе I1 только один, дерево гаплотипов гаплогруппы столь симметричное. Такое встречается очень редко. ID00020 – потомок этого общего предка.
Приложение. Здесь приводятся детальные геномные данные по составу снипов, обнаруженных у носителей гаплотипов с ветви ID00020. У него должны быть практически такие, за исключением, возможно, терминальных снипов. Но, возможно, и они те же, как в нескольких приведенных случаях. Выделены снипы от М223 и ниже:
Kit No. 38184 (Matthew Hamilton Butcher, американский индеец, но получивший Y-ДНК от европейца, гаплотип 110 на ветви выше): DF29+, M227+, CTS10058+, CTS10140+, CTS10338+, CTS10834+, CTS11036+, CTS11042+, CTS11441+, CTS11526+, CTS11552+, CTS11775+, CTS11783+, CTS11950+, CTS1393+, CTS2193+, CTS2375+, CTS2514+, CTS2524+, CTS2644+, CTS2738+, CTS3517+, CTS3654+, CTS3843+, CTS4088+, CTS4130+, CTS4295+, CTS4437+, CTS4848+, CTS4982+, CTS5167+, CTS5408+, CTS5513+, CTS565+, CTS5650+, CTS5705+, CTS571+, CTS5783+, CTS5884+, CTS5891+, CTS5908+, CTS5993+, CTS6140+, CTS6221+, CTS623+, CTS6265+, CTS6395+, CTS641+, CTS6629+, CTS674+, CTS6932+, CTS7267+, CTS7329+, CTS7831+, CTS7949+, CTS8333+, CTS8345+, CTS8394+, CTS8420+, CTS8716+, CTS88+, CTS8876+, CTS9240+, CTS9264+, CTS9288+, F1209+, F3692+, F719+, L118+, L121+, L123+, L124+, L125+, L132+, L15+, L16+, L187+, L350+, L403+, L41+, L468+, L470+, L498+, L509+, L574+, L575+, L578+, L740+, L748+, L75+, L750+, L751+, L755+, L756+, L758+, L759+, L772+, L80+, L81+, M168+, M170+, M213+, M227+, M235+, M253+, M258+, M294+, M299+, M307+, M89+, M94+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P143+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P30+, P316+, PAGES00026+, PAGES00081+, PAGES00123+, PF2591+, PF2608+, PF2611+, PF2615+, PF2747+, PF2748+, PF2749+, PF2770+, PF3562+, PF3574+, PF3639+, PF3640+, PF3641+, PF3660+, PF3666+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3804+, PF3809+, PF3811+, PF3814+, PF3815+, PF3819+, PF3822+, PF3836+, PF3837+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, YSC0000281+, YSC0000301+, P19+, M42+, M450+, M72-, Z58-, Z63-, Z17694-, S2348-, S4442-, M3453-
Kit No. 38737 (Greathouse, Германия, гаплотип 744): CTS10058+, CTS10140+, CTS10338+, CTS10362+, CTS109+, CTS11036+, CTS11042+, CTS11358+, CTS11441+, CTS11526+, CTS11552+, CTS11575+, CTS11726+, CTS11775+, CTS11783+, CTS11950+, CTS125+, CTS12632+, CTS1393+, CTS1996+, CTS2193+, CTS2375+, CTS2514+, CTS2524+, CTS2536+, CTS2644+, CTS2738+, CTS3331+, CTS3431+, CTS3517+, CTS3536+, CTS3654+, CTS3662+, CTS3843+, CTS3868+, CTS3996+, CTS4088+, CTS4130+, CTS4295+, CTS4364+, CTS4368+, CTS4443+, CTS4740+, CTS4848+, CTS4982+, CTS5167+, CTS5318+, CTS5408+, CTS5457+, CTS5513+, CTS5532+, CTS565+, CTS5650+, CTS5705+, CTS571+, CTS5783+, CTS5891+, CTS5908+, CTS5993+, CTS6135+, CTS6140+, CTS6221+, CTS6265+, CTS6383+, CTS6395+, CTS641+, CTS6629+, CTS674+, CTS6800+, CTS6907+, CTS6932+, CTS7267+, CTS7329+, CTS7502+, CTS7831+, CTS7922+, CTS7933+, CTS7949+, CTS8243+, CTS8333+, CTS8345+, CTS8394+, CTS8420+, CTS8716+, CTS88+, CTS8876+, CTS8980+, CTS9240+, CTS9264+, CTS9288+, CTS9828+, F1046+, F1209+, F1302+, F1320+, F1329+, F1450+, F1460+, F1704+, F1714+, F1753+, F1767+, F2048+, F2075+, F2142+, F2155+, F2302+, F2345+, F2366+, F2402+, F2408+, F2587+, F2688+, F2710+, F2794+, F2837+, F2985+, F2993+, F3033+, F3111+, F3136+, F3335+, F3368+, F3402+, F3556+, F3692+, F4188+, F719+, F922+, L118+, L121+, L124+, L125+, L132+, L15+, L157+, L16+, L187+, L350+, L403+, L468+, L470+, L498+, L509+, L574+, L575+, L578+, L740+, L748+, L75+, L750+, L751+, L755+, L756+, L758+, L759+, L772+, L80+, M139+, M168+, M170+, M235+, M253+, M294+, M307+, M42+, M450+, M89+, M94+, P123+, P124+, P126+, P127+, P130+, P135+, P136+, P138+, P14+, P141+, P145+, P146+, P148+, P151+, P158+, P159+, P160+, P166+, P187+, P30+, P305+, PAGES00026+, PAGES00081+, PAGES00123+, PF1016+, PF1029+, PF1031+, PF1040+, PF1046+, PF1061+, PF1092+, PF1097+, PF110+, PF1203+, PF1269+, PF1276+, PF192+, PF210+, PF212+, PF223+, PF234+, PF258+, PF2591+, PF2593+, PF2599+, PF2608+, PF2611+, PF2615+, PF2624+, PF263+, PF2643+, PF272+, PF2745+, PF2747+, PF2748+, PF2749+, PF2770+, PF278+, PF292+, PF316+, PF325+, PF342+, PF3515+, PF3517+, PF3518+, PF3534+, PF3560+, PF3561+, PF3562+, PF3574+, PF3578+, PF3586+, PF3588+, PF3590+, PF3594+, PF3596+, PF3600+, PF3604+, PF3605+, PF3611+, PF3612+, PF3616+, PF3618+, PF3625+, PF3639+, PF3640+, PF3641+, PF3642+, PF3654+, PF3660+, PF3666+, PF3672+, PF3675+, PF3677+, PF3686+, PF3694+, PF3780+, PF3800+, PF3804+, PF3806+, PF3807+, PF3809+, PF3811+, PF3814+, PF3815+, PF3819+, PF3822+, PF3836+, PF3837+, PF500+, PF667+, PF719+, PF725+, PF779+, PF796+, PF803+, PF815+, PF821+, PF840+, PF844+, PF892+, PF937+, PF951+, PF954+, PF970+, V168+, V186+, V189+, V205+, V221+, V241+, V250+, V52+, V9+, YSC0000056+, YSC0000256+, YSC0000257+, YSC0000259+, YSC0000260+, YSC0000264+, YSC0000265+, YSC0000267+, YSC0000272+, YSC0000280+, YSC0000281+, YSC0000298+, YSC0000299+, YSC0000300+, YSC0000301+, Z138+, Z139+, M170+, M258+, M307+, P19+, P30+, P38+, P109-, P259-, P37-, M161-, M72-, M26-, M21-, M223-, M227-
Kit No. 152077 (Jens Peter Nielsen, Дания, гаплотип 745): CTS10058+, CTS10140+, CTS10338+, CTS10362+, CTS109+, CTS11036+, CTS11042+, CTS11358+, CTS11441+, CTS11526+, CTS11552+, CTS11575+, CTS11726+, CTS11775+, CTS11783+, CTS11950+, CTS125+, CTS12632+, CTS1393+, CTS1996+, CTS2193+, CTS2375+, CTS2514+, CTS2524+, CTS2536+, CTS2644+, CTS2738+, CTS3331+, CTS3431+, CTS3517+, CTS3536+, CTS3654+, CTS3662+, CTS3843+, CTS3868+, CTS3996+, CTS4088+, CTS4130+, CTS4295+, CTS4364+, CTS4368+, CTS4443+, CTS4740+, CTS4848+, CTS4982+, CTS5167+, CTS5318+, CTS5408+, CTS5457+, CTS5513+, CTS5532+, CTS565+, CTS5650+, CTS5705+, CTS571+, CTS5783+, CTS5891+, CTS5908+, CTS5993+, CTS6135+, CTS6140+, CTS6221+, CTS6265+, CTS6383+, CTS6395+, CTS641+, CTS6629+, CTS674+, CTS6800+, CTS6907+, CTS6932+, CTS7267+, CTS7329+, CTS7502+, CTS7831+, CTS7922+, CTS7933+, CTS7949+, CTS8243+, CTS8333+, CTS8345+, CTS8394+, CTS8420+, CTS8716+, CTS88+, CTS8876+, CTS8980+, CTS9240+, CTS9264+, CTS9288+, CTS9828+, F1046+, F1209+, F1302+, F1320+, F1329+, F1450+, F1460+, F1704+, F1714+, F1753+, F1767+, F2048+, F2075+, F2142+, F2155+, F2302+, F2345+, F2366+, F2402+, F2408+, F2587+, F2688+, F2710+, F2794+, F2837+, F2985+, F2993+, F3033+, F3111+, F3136+, F3335+, F3368+, F3402+, F3556+, F3692+, F4188+, F719+, F922+, L118+, L121+, L124+, L125+, L132+, L15+, L157+, L16+, L187+, L350+, L403+, L468+, L470+, L498+, L509+, L574+, L575+, L578+, L740+, L748+, L75+, L750+, L751+, L755+, L756+, L758+, L759+, L772+, L80+, M139+, M168+, M170+, M235+, M253+, M294+, M307+, M42+, M450+, M89+, M94+, P123+, P124+, P126+, P127+, P130+, P135+, P136+, P138+, P14+, P141+, P145+, P146+, P148+, P151+, P158+, P159+, P160+, P166+, P187+, P30+, P305+, PAGES00026+, PAGES00081+, PAGES00123+, PF1016+, PF1029+, PF1031+, PF1040+, PF1046+, PF1061+, PF1092+, PF1097+, PF110+, PF1203+, PF1269+, PF1276+, PF192+, PF210+, PF212+, PF223+, PF234+, PF258+, PF2591+, PF2593+, PF2599+, PF2608+, PF2611+, PF2615+, PF2624+, PF263+, PF2643+, PF272+, PF2745+, PF2747+, PF2748+, PF2749+, PF2770+, PF278+, PF292+, PF316+, PF325+, PF342+, PF3515+, PF3517+, PF3518+, PF3534+, PF3560+, PF3561+, PF3562+, PF3574+, PF3578+, PF3586+, PF3588+, PF3590+, PF3594+, PF3596+, PF3600+, PF3604+, PF3605+, PF3611+, PF3612+, PF3616+, PF3618+, PF3625+, PF3639+, PF3640+, PF3641+, PF3642+, PF3654+, PF3660+, PF3666+, PF3672+, PF3675+, PF3677+, PF3686+, PF3694+, PF3780+, PF3800+, PF3804+, PF3806+, PF3807+, PF3809+, PF3811+, PF3814+, PF3815+, PF3819+, PF3822+, PF3836+, PF3837+, PF500+, PF667+, PF719+, PF725+, PF779+, PF796+, PF803+, PF815+, PF821+, PF840+, PF844+, PF892+, PF937+, PF951+, PF954+, PF970+, V168+, V186+, V189+, V205+, V221+, V241+, V250+, V52+, V9+, YSC0000056+, YSC0000256+, YSC0000257+, YSC0000259+, YSC0000260+, YSC0000264+, YSC0000265+, YSC0000267+, YSC0000272+, YSC0000280+, YSC0000281+, YSC0000298+, YSC0000299+, YSC0000300+, YSC0000301+, Z138+, Z139+, M170+, M258+, M307+, P19+, P30+, P38+, P109-, P259-, P37-, M161-, M72-, M26-, M21-, M223-, M227-
Kit N10060 (Robert Wade, Англия, гаплотип 57): M170+, M253+, M258+, M307+, P19+, P30+, P38+, P109-, P259-, P37-, M161-, M72-, M26-, M21-, M223-, M227-
Эту гаплогруппу имели два человека, один субклада I2a1-L147.2, второй – I2a2-M223, с 67- и 111-маркерным гаплотипом, соответственно.
KLIN ID00004
Представлен 67-маркерный гаплотип, сообщено, что гаплогруппа I2a.
13 24 17 11 14 15 11 13 12 13 11 30 – 17 8 10 11 11 25 15 20 0 12 14 15 15 – 10 10 20 21 15 12 19 17 35 35 11 10 – 11 8 15 15 7 12 10 8 11 9 12 22 22 16 10 12 12 12 7 10 30 21 13 14 10 13 11 11 12 9
Эта гаплогруппа распространена по всей Восточной Европе, а именно в странах – Греция, Сербия, Босния-Герцеговина, Македония, Чехия, Словакия, Польша, Болгария, Белоруссия, Россия, Украина, Венгрия, а также немного в Германии, Румынии и Италии, и у подавляющего большинства тестированных был обнаружен снип I2a-CTS10228, он же L147.2 (см. диаграмму ISOGG ниже; справа – датировка образования снипа по данным компании YFull). Это – типичный снип Восточной Европы. По виду гаплотипа совершенно ясно, что он есть и у заказчика. Вопрос – сможем ли мы по виду гаплотипа предсказать некоторые нижестоящие снипы?

В базе данных (Проект FTDNA I2a имеются 245 гаплотипов в 67-маркерном формате. На дереве, построенном из всех этих гаплотипов, гаплотип ID00004 находится справа (помечен символом Х), он относится к родственной большой ветви, которая занимает большую часть правой, компактной ветви дерева. Компактная – это значит относительно молодая ветвь, по сравнению с более рыхлыми ветвями. Иначе говоря, компактная ветвь свидетельствует о ее относительно недавнем происхождении. Расчеты показывают, что этой ветви примерно 2300 лет, то есть она образовалась в конце прошлой эры. Практически все гаплотипы этой ветви сопряжены со снипом CTS10228/L147.2. Но у нее есть и нисходящие снипы, например
L147.2 > YP204/S17250 >Z16971
L147.2 > YP204/S17250 >Y4882 > A811
L147.2 > YP204/S17250 >Y4882 > A1328
L147.2 > YP204/S17250 >A356/Z16983
L147.2 > Y4460 > Y3118
L147.2 > Z17855
которые в 67-маркерных гаплотипах на дереве не разделяются. Точнее, возможно, они могут расходиться по разным частям ветви, но слишком мало носителей гаплотипов в Проекте определяли у себя столь глубокие снипы. Их будет определять московская Лаборатория ДНК-генеалогии.
Есть еще одна причина, почему перечисленные субклады не разделяются на дереве гаплотипов – практически все они образовались почти одновременно (примерно 2100 лет назад) с родительским снипом CTS10228 (примерно 2300 лет назад). Так что их не разделяет не только 67-маркерное дерево, но и 111-маркерное дерево этой отдельной ветви:
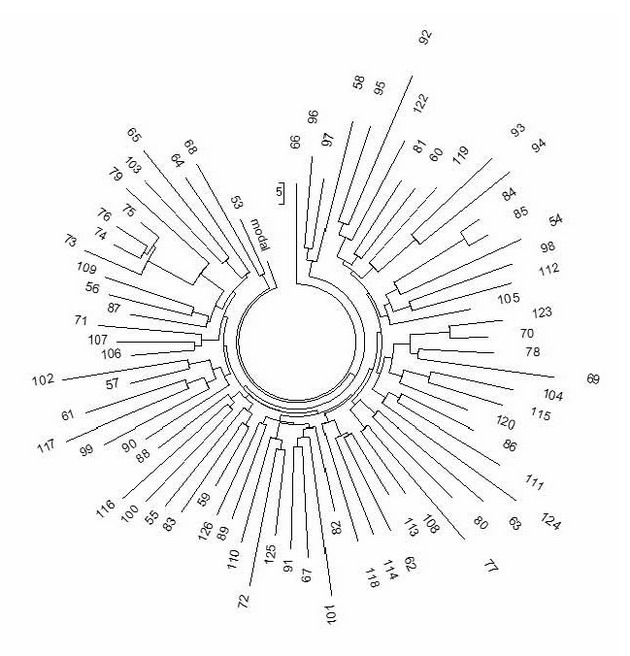
Мы видим, что и на 111-маркерном дереве нет выраженных ветвей, которые можно было бы отнести к конкретным снипам. Базовый гаплотип ветви в 67-маркерном формате:
13 24 16 11 14 15 11 13 13 13 11 31 – 17 8 10 11 11 25 15 20 32 12 14 15 15 – 10 10 21 21 15 12 18 18 34 35 11 10 – 11 8 15 15 7 12 10 8 11 9 12 22 22 16 10 12 12 12 7 10 30 21 13 14 10 13 11 11 12 9
(выделены 8 мутаций от представленного гаплотипа). Это означает, что гаплотип удален от предкового на 8/0.12 = 67 → 72 условных поколения, то есть примерно на 1800 лет. Это, конечно, примерно, так как по двум гаплотипам точных расчетов не бывает – одна случайная мутация в представленном гаплотипе сдвинет расчетное расстояние больше, чем на 200 лет. В любом случае ясно, что предки заказчика вели ДНК-линию ID00004 практически напрямую от общего предка данной ветви.
История гаплогруппы/субклада I2a-L147.2 c возрастом примерно 2300 лет неоднократно обсуждалась на Переформате. Гаплогруппу I2a находили в захоронениях в центральной Европе с датировками 7000-5000 лет назад, затем примерно 4500 лет назад она из центральной Европы исчезла, и разошлась на две ветви. Одна (точнее, несколько ветвей, см. первую диаграмму выше) передвинулась (или бежала) на Британские острова, и общие предки этой совокупности ветвей уходят примерно на 4500-5000 лет назад. Другая ветвь субклада I2a-L147.2, прошла в Европе бутылочное горлышко популяции, возродилась около 2300 лет назад, на Дунае-Карпатах, и разошлась по всей Восточной Европе в первой половине I тыс. нашей эры. Возможно, это ее историки и археологи приняли за «происхождение славян», и это о ней идет речь в древних летописях, таких как Повесть временных лет. Если ПВЛ права, то славяне гаплогруппы I2a-L147.2 вышли из Норика, в восточных Альпах, к северу от северной части Адриатического моря. Тавриски, жители Норика, известны со второй половины I тыс. до н.э.
Если бы ID00004 сделал себе тест BigY (или другой геномный тест на Y-хромосому), то результат бы близок (или идентичен) следующему (здесь выделены вышестоящие снипы субклада I2a-CTS10228/L147.2). Остальные отмеченные снипы относятся к еще более вышестоящим гаплотипам, как, например, M42 – сводная гаплогруппа ВТ; V9, V52 и V189 – это всё сводная гаплогруппа СТ; М89 – гаплогруппа F, L15 и L16 – сводная гаплогруппа IJK, L41 – гаплогруппа I, и так далее. Как видим, единственное полезное, что дал здесь тест BigY (за 599 долларов) – это идентификация самого нижнего снипа I2a-CTS10228. Более нижестоящих снипов здесь нет, а приведенный выше снип Z17855 в списке ниже отрицательный (четвертый от конца).
Приведенный ниже набор снипов взят из открытой базы данных, он принадлежит потомку польского князя Святополк-Четвертинского, по легендам – из ополячившихся Рюриковичей. Кстати, князь Борис Антонович Четвертинский (1784-1865), участник наполеоновский войн, полковник русской армии – из того же польского рода. Большинство этих снипов, если не все, должны быть найдены и у ID00004. Впрочем, все – это только если глубокий субклад окажется точно таким же, как и у Четвертинского. Но интересно, что глубоких снипов ниже L147.2 у Четвертинского не обнаружено, самый последний, терминальный, это CTS10228, то есть L147.2.
CTS4002+, CTS4039+, CTS4088+, CTS410+, CTS4437+, CTS4848+, CTS4982+, CTS5044+, CTS5375+, CTS5650+, CTS5727+, CTS5884+, CTS5908+, CTS5966+, CTS5985+, CTS623+, CTS6265+, CTS674+, CTS6932+, CTS7175+, CTS7213+, CTS7218+, CTS7329+, CTS7831+, CTS8239+, CTS8333+, CTS8345+, CTS8420+, CTS8486+, CTS88+, CTS8876+, CTS9240+, CTS9264+, CTS9349+, CTS10058+, CTS10228+, CTS10834+, CTS10936+, CTS11030+, CTS11441+, CTS11768+, CTS1293+, CTS176+, CTS1846+, CTS2193+, CTS2514+, CTS3296+, CTS3517+, CTS3654+, F1209+, F3145+, F3692+, F719+, L132+, L15+, L16+, L178+, L350+, L403+, L41+, L460+, L468+, L470+, L498+, L578+, M42+, M423+, M438+, M89+, M94+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P143+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P316+, P38+, PAGES00026+, PAGES00081+, PF2591+, PF2608+, PF2611+, PF2615+, PF2747+, PF2748+, PF2749+, PF2770+, PF3562+, PF3574+, PF3626+, PF3638+, PF3639+, PF3640+, PF3641+, PF3644+, PF3651+, PF3652+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3801+, PF3802+, PF3804+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF3966+, PF4058+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, L621+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, M168+, M170+, M213+, M235+, M258+, M294+, M299+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, M423+, S7721+, V19-, P41-, Z17694-, Z2103-, Z2109-, M3453-, S2348-, S4442-, P61-, L584-, CTS3802-, DF41-
Поскольку речь зашла о Рюриковичах, напомним, что у группы князей Российского дворянского собрания – гаплогруппа N1c1, у Четвертинского (и у заказчика) – гаплогруппа I2a. Рюрик, если такой вобще существовал, мог иметь только одну гаплогруппу. Так что вопрос о многочисленных Рюриковичах пока остается открытым, если у всех у них есть документальные основания утверждать, что они Рюриковичи. Насколько мне известно, документальных оснований ни у кого нет, но есть устоявшиеся мнения специалистов по княжеским генеалогиям. Причем наверняка у польских генеалогов мнение одно, а у российских – мнение другое. Пусть они между собой и разбираются, но ДНК-генеалогия предоставляет им новые данные для расмотрения.
Возможно, московская Лаборатория ДНК-генеалогии сможет идентифицировать у ID00004 нижестоящие (по отношению к CTS1028) снипы, и понять, в каких регионах они преимущественно выражены. Тогда персональную генеалогию можно будет еще далее углубить.
KLIN ID00019
Представлен 111-маркерный гаплотип, сообщено, что гаплогруппа I2b, субклад I2b1, снип М223.
15 23 15 10 15 15 11 14 12 14 12 32 16 8 9 11 11 25 14 20 28 11 14 14 15 11 10 19 21 15 14 18 21 32 40 12 10 11 8 16 16 8 11 10 8 10 9 12 22 22 15 11 12 12 15 9 14 27 21 11 13 12 12 11 12 12 11 32 13 8 15 10 24 27 16 12 11 14 11 12 9 13 11 10 10 12 32 10 12 22 13 11 10 20 15 23 9 24 14 12 14 27 12 21 18 12 15 17 9 12 11
В представленных данных использована устаревшая номенклатура 2010 года, с тех пор снип М223 относят к субкладу I2a2a (см. диаграмму ниже, в сокращенном виде). В правой колонке представлены (в годах, лет назад) примерные датировки возникновения древнейших и некоторых других снипов (которые здесь представляют для нас интерес), рассчитанные компанией YFull по геномным данным.

Сиреневым цветом выше обозначен субклад I2a-L147.2, наиболее распространенный в гаплогруппе I в Восточной Европе – от Греции до Прибалтики, включая Россию, Украину, Белоруссию, страны бывшей Югославии; красной строкой выделен субклад представленного гаплотипа, I2a-M223. Сам по себе субклад древний, ему 17400 лет, и, конечно, у него имеются нижестоящие суклады, к выявлению которых мы в настоящей Интерпретации приблизимся. Желтым цветом выделены субклады, которые могут быть у ID00019, как будет показано ниже.
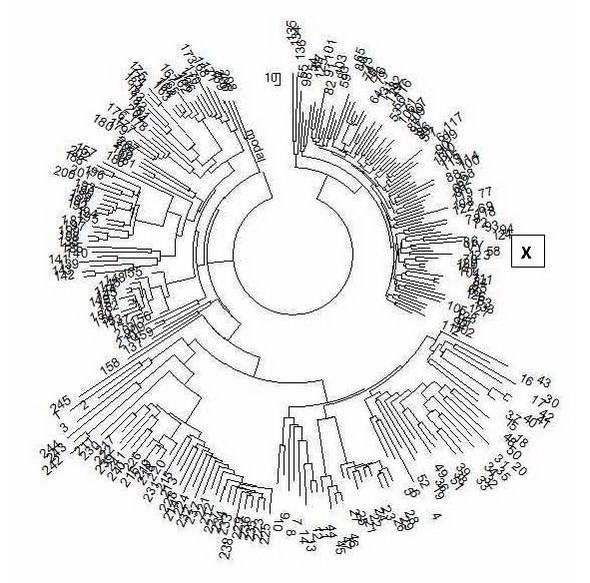
Попытаемся определить место представленного гаплотипа на общем дереве 111-маркерных гаплотипов гаплогруппы I2 из Проекта I FTDNA. Для этого возьмем все 63 111-маркерных гаплотипов гаплогруппы I2 Проекта, добавим к ним представленный гаплотип ID00019, и построим дерево из полученных 64 гаплотипов с помощью профессиональной компьютерной программы (PHYLIP). Цель этого эксперимента – выяснить, в окружении каких гаплотипов и из каких субкладов окажется представленный гаплотип. Дерево этих 111-маркерных гаплотипов гаплогруппы I2 приведено ниже.
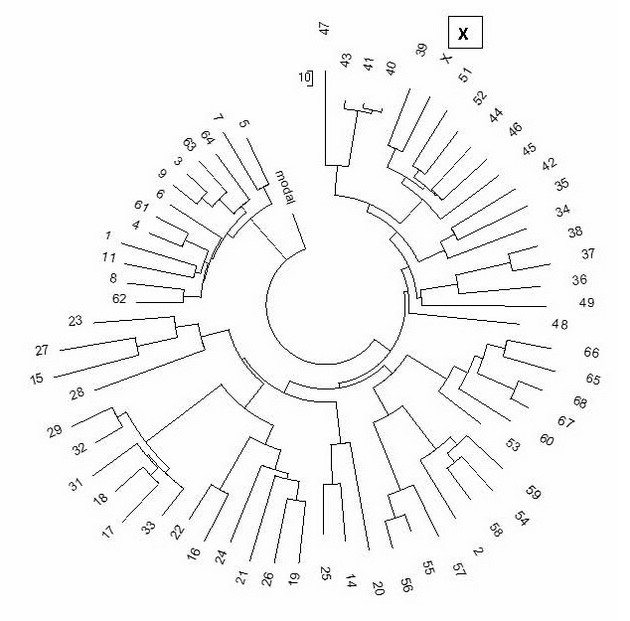
Положение представленного гаплотипа обозначено на дереве индексом Х. Это – отдельная, хорошо выраженная ветвь из семи подобных по виду, предположительно родственных гаплотипов. Для сведения, базовый (предковый) гаплотип всего дерева I2 следующий
13 24 16 10 13 15 11 13 12 13 11 30 17 8 10 11 11 25 15 20 29 12 14 15 15 10 10 19 20 15 14 17 18 34 36 12 10 11 8 15 16 8 11 10 8 11 9 11 21 22 16 11 12 12 15 8 13 25 21 11 13 11 13 11 12 12 11 30 14 8 15 11 25 27 18 12 11 12 12 12 9 12 11 10 11 12 31 11 12 22 14 11 10 22 15 20 11 23 16 11 15 25 12 22 18 12 14 17 9 12 11
Представленный гаплотип отличается от него на 70 мутаций. Это эквивалентно 70/0.198 = 354 → 537 условных поколений, или примерно 13425 лет между ними (0.198 – это константа скорости мутации для 111-маркерных гаплотипов, стрелка – поправка на возвратные мутации). Иначе говоря, общий предок гаплогруппы чрезвычайно удален по времени от ID00019.
Все 64 гаплотипа имеют 3906 мутаций от базового гаплотипа, это соответствует времени, когда жил общий предок современных носителей этого гаплотипа, и которое составляет в данном случае 3906/64/0.198 = 308 → 440 условных поколений (по 25 лет каждое, под этот срок калибровались константы скорости мутаций), или 11000±1100 лет назад. Ясно, что он жил намного позже времени образования субклада I2 (примерно 27500 лет назад, определено по снипам компанией YFull), и это свидетельствует, что гаплогруппа I2 прошла около 11000 лет назад жесткое бутылочное горлышко популяции, возможно, связанной с периодом оледенения в Европе. Общий предок заказчика и гаплогруппы I2 жил примерно (13425+11000)/2 = 12200 лет назад, то есть еще до прохождения бутылочного горлышка.
Мы видим, что представленный гаплотип находится на одной ветви с гаплотипами под номерами 39, 44-46, 51, 52. Базовый гаплотип этой ветви
15 23 15 10 15 15 11 13 12 14 12 32 15 8 10 11 11 25 14 20 28 11 14 14 15 11 10 19 21 14 14 17 19 33 39 12 10 11 8 15 16 8 11 10 8 10 9 12 21 22 15 11 12 12 14 9 13 27 20 11 13 12 12 11 12 12 11 31 13 8 15 11 24 27 16 12 11 13 11 13 9 12 11 10 11 12 32 10 12 22 13 11 10 20 15 22 9 23 14 12 14 28 12 21 18 12 15 17 9 12 11
отличается на 23 мутации (отмечены) от представленного гаплотипа, то есть гаплотип удален от предкового для данной ветви на 23/0.198 = 116 → 132 условных поколения (по 25 лет), то есть примерно на 3300 лет. В то же время все семь гаплотипов ветви имеют 163 мутации от показанного выше базового гаплотипа, то есть общий предок этой ветви жил 163/7/0.198 = 118 → 134 условных поколения, то есть 3350±425 лет назад. Мы видим, что эти две величины совпадают практически идеально (3300 и 3350 лет, тем более в пределах погрешности расчетов). Это означает, что представленный гаплотип почти идеально вписывается в данную ветвь из семи гаплотипов, и соответствует ей и по гаплотипу, и по удалению от нее, и по расчитанному возрасту общего предка ветви. Так что ветвь гаплотипа ID00019 мы нашли.
Посмотрим, к каким субкладам относятся родственные (в ДНК-генеалогическом смысле) гаплотипы, под номерами 39, 44, 45, 46, 51 и 52. Ближе всего на дереве к представленному гаплотипу сидит гаплотип 39 (Англия). У него снип Р95, с возрастом 3100 лет (выделен желтым цветом на диаграмме в начале этого материала). Это практически совпадает с возрастом рассматриваемой ветви, 3350±425 лет. Далее идут гаплотипы 51 и 52 (Шотландия и Швеция), они не типированы на снипы глубже М223, как и у ID00019. Но проект определяет их к снипам не выше Z161, L801 и CTS6433. Последние три гаплотипа на ветви, 44, 45 и 46 (Англия, Португалия и Швейцария), имеют снипы соответственно Z79, CTS6433 и CTS6433, с возрастом 2400, 3800 и 3800 лет. Таким образом, и ветвь ID00019 относительно молодая, и снипы на ней по возрасту соответствуют, учитывая, что и возраст снипов определен с определенной погрешностью.
Подводя итоги, следует заключить, что снипы ID00019 должны быть в ареале снипов, обозначенных желтым цветом на диаграмме снипов в начале Интерпретации, и скорее всего среди снипов P95, CTS6433 и Z79. Московская лаборатория ДНК-генеалогии планирует определять все эти снипы.
Исторический путь к представленному гаплотипу примерно следующий. Гаплогруппа I образовалась около 43 тысяч лет назад (из сводной гаплогруппы IJ), около 28 тысяч лет она разошлась на гаплогруппы I1 и I2, и из последней (как и из первой) образовалось множество нижестоящих субкладов. Дальнейшая история гаплогруппы I2 описана в предшествующих Интерпретациях; в итоге большинство носителей гаплогруппы I2a из Восточной Европы относятся к нисходящим ветвям субклада L147.2, общий предок которых жил в конце I тыс. до н.э., примерно 2300 лет назад. ID00029 – представитель другой, параллельной ветви, М223, но относительно недавних субкладов, общий предок которых жил во второй половине II тыс. до н.э., примерно 3300 лет назад.
Приложение. Здесь приводятся детальные геномные данные по составу снипов, обнаруженных у носителей гаплотипов с ветви ID00029. У него должны быть практически такие, за исключением, возможно, терминальных снипов. Но, возможно, и они те же, как в нескольких приведенных случаях. Выделены снипы от М223 и ниже.
Kit No. 227059 (Англия, гаплотип 39 на ветви): CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10362+, CTS109+, CTS11358+, CTS11441+, CTS11575+, CTS11726+, CTS125+, CTS12632+, CTS1977+, CTS1996+, CTS2193+, CTS2392+, CTS2514+, CTS2536+, CTS3296+, CTS3326+, CTS3331+, CTS3431+, CTS3517+, CTS3536+, CTS3654+, CTS3662+, CTS3868+, CTS3996+, CTS4039+, CTS4088+, CTS429+, CTS4314+, CTS4348+, CTS4364+, CTS4368+, CTS4443+, CTS4740+, CTS4848+, CTS4982+, CTS5286+, CTS5318+, CTS5457+, CTS5532+, CTS5650+, CTS5727+, CTS5908+, CTS6135+, CTS6136+, CTS616+, CTS6265+, CTS6331+, CTS6383+, CTS674+, CTS6800+, CTS6907+, CTS6932+, CTS7329+, CTS7331+, CTS7502+, CTS7682+, CTS7831+, CTS7865+, CTS7922+, CTS7933+, CTS7934+, CTS8243+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS8980+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, CTS9828+, F1046+, F1209+, F1302+, F1320+, F1329+, F1450+, F1460+, F1704+, F1714+, F1753+, F1767+, F2048+, F2075+, F2142+, F2155+, F2302+, F2345+, F2366+, F2402+, F2587+, F2688+, F2710+, F2794+, F2837+, F2985+, F2993+, F3111+, F3136+, F3335+, F3368+, F3402+, F3556+, F3692+, F4188+, F719+, F922+, L132+, L15+, L16+, L34+, L35+, L350+, L37+, L403+, L460+, L468+, L470+, L498+, L578+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M139+, M168+, M170+, M223+, M235+, M294+, M42+, M89+, M94+, P123+, P124+, P126+, P127+, P130+, P135+, P136+, P138+, P14+, P141+, P145+, P146+, P148+, P151+, P158+, P159+, P160+, P166+, P187+, P214+, P215+, P216+, P217+, P219+, P220+, P221+, P222+, P223+, P305+, P95+, PAGES00026+, PAGES00081+, PF1016+, PF1029+, PF1031+, PF1040+, PF1046+, PF1061+, PF1092+, PF1097+, PF110+, PF1203+, PF1269+, PF1276+, PF192+, PF210+, PF212+, PF223+, PF234+, PF258+, PF2591+, PF2593+, PF2599+, PF2608+, PF2611+, PF2615+, PF2624+, PF263+, PF2643+, PF272+, PF2745+, PF2747+, PF2748+, PF2749+, PF2770+, PF278+, PF292+, PF316+, PF325+, PF342+, PF3515+, PF3517+, PF3518+, PF3534+, PF3560+, PF3561+, PF3562+, PF3573+, PF3574+, PF3578+, PF3586+, PF3588+, PF3590+, PF3594+, PF3595+, PF3596+, PF3600+, PF3604+, PF3605+, PF3607+, PF3611+, PF3612+, PF3616+, PF3618+, PF3621+, PF3623+, PF3625+, PF3626+, PF3634+, PF3639+, PF3640+, PF3641+, PF3642+, PF3644+, PF3651+, PF3652+, PF3654+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3686+, PF3694+, PF3780+, PF3798+, PF3800+, PF3801+, PF3802+, PF3804+, PF3806+, PF3807+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF500+, PF667+, PF719+, PF725+, PF779+, PF796+, PF803+, PF815+, PF821+, PF840+, PF844+, PF892+, PF937+, PF951+, PF954+, PF970+, V168+, V186+, V189+, V205+, V221+, V241+, V250+, V52+, V9+, YSC0000056+, YSC0000256+, YSC0000260+, YSC0000265+, YSC0000267+, YSC0000272+, YSC0000280+, YSC0000281+, YSC0000298+, YSC0000300+, Z161+, Z162+, Z163+, Z164+, Z165+, Z168+, Z170+, Z172+, Z174+, Z175+, Z176+, Z177+, Z178+, Z179+, Z181+, Z183+, Z184+, Z186+, Z188+, Z189+, Z77+, CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10834+, CTS11441+, CTS1977+, CTS2193+, CTS2392+, CTS2514+, CTS3296+, CTS3326+, CTS3517+, CTS3654+, CTS4039+, CTS4088+, CTS429+, CTS4348+, CTS4437+, CTS4848+, CTS4982+, CTS5286+, CTS5650+, CTS5727+, CTS5884+, CTS5908+, CTS6136+, CTS616+, CTS623+, CTS6265+, CTS6331+, CTS674+, CTS6932+, CTS7329+, CTS7331+, CTS7682+, CTS7831+, CTS7865+, CTS7934+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, F1209+, F3692+, F719+, L132+, L15+, L16+, L181+, L34+, L35+, L350+, L36+, L37+, L403+, L41+, L460+, L468+, L470+, L498+, L578+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M168+, M170+, M213+, M223+, M235+, M258+, M294+, M299+, M42+, M438+, M89+, M94+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P143+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P214+, P216+, P217+, P218+, P219+, P220+, P221+, P222+, P223+, P316+, P38+, P95+, PAGES00026+, PAGES00081+, PF2591+, PF2608+, PF2611+, PF2615+, PF2747+, PF2748+, PF2749+, PF2770+, PF3562+, PF3574+, PF3626+, PF3639+, PF3640+, PF3641+, PF3644+, PF3651+, PF3652+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3801+, PF3802+, PF3804+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, YSC0000281+, Z161+, Z163+, Z164+, Z165+, Z168+, Z170+, Z174+, Z175+, Z176+, Z178+, Z179+, Z184+, Z186+, Z188+, P37-, S2348-, S4442-
Kit No. 173448 (Англия, гаплотип 44): Z77+, L800+, L801+, V218+, Z186+, Z78+, Z79+, L1198+, Z190+, Z185+, Z171+, Z166+, CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10834+, CTS11441+, CTS2134+, CTS2193+, CTS2392+, CTS2514+, CTS3296+, CTS3326+, CTS3517+, CTS3654+, CTS4039+, CTS4088+, CTS429+, CTS4348+, CTS4437+, CTS4848+, CTS4982+, CTS5286+, CTS5650+, CTS5727+, CTS5884+, CTS5908+, CTS6136+, CTS616+, CTS623+, CTS6265+, CTS6331+, CTS6433+, CTS674+, CTS6932+, CTS7329+, CTS7331+, CTS7682+, CTS7831+, CTS7865+, CTS7934+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, F1209+, F3692+, F719+, L104+, L132+, L15+, L16+, L181+, L34+, L35+, L350+, L36+, L37+, L403+, L41+, L460+, L468+, L498+, L578+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M168+, M170+, M213+, M223+, M235+, M258+, M294+, M299+, M42+, M438+, M89+, M94+, P108+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P143+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P214+, P216+, P217+, P218+, P219+, P220+, P221+, P222+, P223+, P305+, P316+, PAGES00026+, PAGES00081+, PF2591+, PF2608+, PF2611+, PF2615+, PF2624+, PF2747+, PF2748+, PF2749+, PF2770+, PF3562+, PF3574+, PF3626+, PF3639+, PF3640+, PF3641+, PF3644+, PF3651+, PF3652+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3801+, PF3802+, PF3804+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, PK1+, SRY10831+, V221+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, Z163+, Z164+, Z165+, Z168+, Z170+, Z174+, Z175+, Z178+, Z179+, Z184+, Z185+, Z188+, Z190+, Z79+, L707+, M223+, M253-, M26-, M284-, M379-, P78-, P95-, S2348-, S4442-
Kit No. 18187 (Португалия, гаплотип 45): CTS674+, CTS6800+, CTS6907+, CTS6932+, CTS7329+, CTS7331+, CTS7502+, CTS7682+, CTS7831+, CTS7865+, CTS7922+, CTS7933+, CTS7934+, CTS8243+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS8980+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, CTS9828+, F1046+, F1209+, F1302+, F1320+, F1329+, F1450+, F1460+, F1704+, F1714+, F1753+, F1767+, F2048+, F2075+, F2142+, F2155+, F2302+, F2345+, F2366+, F2402+, F2587+, F2688+, F2710+, F2794+, F2837+, F2985+, F2993+, F3111+, F3136+, F3335+, F3368+, F3402+, F3556+, F3692+, F4188+, F719+, F922+, L132+, L15+, L16+, L34+, L35+, L350+, L37+, L403+, L41+, L460+, L468+, L470+, L498+, L578+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M139+, M168+, M170+, M223+, M235+, M294+, M42+, M89+, M94+, P123+, P124+, P126+, P127+, P130+, P135+, P136+, P138+, P14+, P141+, P145+, P146+, P148+, P151+, P158+, P159+, P160+, P166+, P187+, P214+, P215+, P216+, P217+, P219+, P220+, P221+, P222+, P223+, PF1016+, PF1029+, PF1031+, PF1040+, PF1046+, PF1061+, PF1092+, PF1097+, PF110+, PF1203+, PF1269+, PF1276+, PF192+, PF210+, PF212+, PF223+, PF234+, PF258+, PF2591+, PF2593+, PF2599+, PF2608+, PF2611+, PF2615+, PF2624+, PF263+, PF2643+, PF272+, PF2745+, PF2747+, PF2748+, PF2749+, PF2770+, PF278+, PF292+, PF316+, PF325+, PF342+, PF3515+, PF3517+, PF3518+, PF3534+, PF3560+, PF3561+, PF3562+, PF3573+, PF3574+, PF3578+, PF3586+, PF3588+, PF3590+, PF3594+, PF3595+, PF3596+, PF3600+, PF3604+, PF3605+, PF3607+, PF3611+, PF3612+, PF3616+, PF3618+, PF3621+, PF3623+, PF3625+, PF3626+, PF3634+, PF3639+, PF3640+, PF3641+, PF3642+, PF3644+, PF3651+, PF3652+, PF3654+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3686+, PF3694+, PF3780+, PF3798+, PF3800+, PF3801+, PF3802+, PF3804+, PF3806+, PF3807+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF500+, PF667+, PF719+, PF725+, PF779+, PF796+, PF803+, PF815+, PF821+, PF840+, PF844+, PF892+, PF937+, PF951+, PF954+, PF970+, V186+, V189+, V205+, V52+, V9+, YSC0000056+, YSC0000256+, YSC0000260+, YSC0000265+, YSC0000267+, YSC0000272+, YSC0000280+, YSC0000281+, YSC0000298+, YSC0000300+, Z161+, Z162+, Z163+, Z164+, Z165+, M170+, M223+, M258+, P38+, L801+, Z186+, Z76+, L1316+, CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10362+, CTS109+, CTS11358+, CTS11441+, CTS11575+, CTS11726+, CTS125+, CTS12632+, CTS1996+, CTS2193+, CTS2392+, CTS2514+, CTS2536+, CTS3296+, CTS3326+, CTS3331+, CTS3431+, CTS3517+, CTS3536+, CTS3654+, CTS3662+, CTS3868+, CTS3996+, CTS4039+, CTS4088+, CTS429+, CTS4314+, CTS4348+, CTS4364+, CTS4368+, CTS4443+, CTS4740+, CTS4848+, CTS4982+, CTS5286+, CTS5318+, CTS5457+, CTS5532+, CTS5650+, CTS5727+, CTS5908+, CTS6135+, CTS6136+, CTS616+, CTS6265+, CTS6331+, CTS6383+, CTS6433+, F3692+, F719+, L104+, L132+, L15+, L16+, L181+, L34+, L35+, L350+, L36+, L37+, L403+, L41+, L460+, L468+, L498+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M168+, M170+, M213+, M223+, M235+, M258+, M294+, M299+, M42+, M438+, M89+, M94+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P214+, P216+, P217+, P218+, P219+, P220+, P221+, P222+, P223+, P316+, PAGES00026+, PAGES00081+, PF2591+, PF2608+, PF2611+, PF2615+, PF2747+, PF2748+, PF2749+, PF2770+, L1409+, PF3833+, Z2198+, CTS8444+, CTS6497+, CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10834+, CTS11441+, CTS2134+, CTS2193+, CTS2392+, CTS2514+, CTS3296+, CTS3326+, CTS3517+, CTS3654+, CTS4039+, CTS4088+, CTS429+, CTS4348+, CTS4437+, CTS4848+, CTS4982+, CTS5286+, CTS5650+, CTS5727+, CTS5884+, CTS5908+, CTS6136+, CTS616+, CTS623+, CTS6265+, CTS6331+, CTS674+, CTS6932+, CTS7329+, CTS7331+, CTS7682+, CTS7831+, CTS7934+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, PF3562+, PF3574+, PF3626+, PF3639+, PF3640+, PF3641+, PF3644+, PF3651+, PF3652+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3801+, PF3802+, PF3804+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, Z172+, Z174+, Z175+, Z176+, Z177+, Z178+, Z179+, P19+, Z181+, Z183+, Z184+, Z186+, V221+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, Z163+, Z164+, Z165+, Z168+, Z170+, Z174+, Z175+, Z178+, Z184+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, Z168+, Z170+, Z188+, Z77+, Z188+, F1209+, F3406-, M161-, Z190-, Z63-, Z79-, L1317-, Z171-, S2348-, S4442-, Z185-, Z187-, L1201-, P30-, P37-, Z180-, PF4225-, DF5-, PF3292-, CTS661-, L1198-, Z190-, Z2059-, Z2084-, Z2074-, L1272-, ZS6-, ZS7-, ZS11-, ZS14-, ZS15-, ZS16-, ZS18-, ZS26-, L1290-, Z78-, Z79-, M284-, M379-, P78-, P95-, L380-, M26-, M307-, M72-, M227-, M253-, M21-, Z166-
Kit No. B3411 (Швейцария, гаплотип 46): L801+, CTS6433+, M223+, L1201-, Z190-, Z78-, CTS5332-
Kit No. 23157 (Шотландия, гаплотип 51): P19+, M170+, M258+, M223+, P19+, P38+, M284-, M379-, P78-, P95-, P30-, P37-, M227-, M253-, M26-, M307-, M72-, M21-, M161-
Kit No. 174472 (Швеция, гаплотип 52): M284-, L126-, M379-, P78-, P95-
Эту гаплогруппу имел один человек, с 67-маркерным гаплотипом.
KLIN ID00005
Сообщено, что субклад I2c-L596, глубже субклад в FTDNA не определяли.
14 24 15 10 12 13 11 13 12 12 11 28 – 18 8 8 11 11 24 15 21 31 11 11 15 15 – 10 10 19 21 15 13 17 17 34 34 14 10 12 8 16 16 8 13 10 8 10 9 12 21 21 16 11 12 13 13 8 14 25 21 12 13 11 13 11 12 12 11
Поскольку глубже, чем I2c-L596, субклад не определяли, то цепочка фактически определенных субкладов в гаплогруппе I довольно короткая: I-M170 > I2-M438 > I2c-L596. Определение в FTDNA проведено правильно, как показывает позиция представленного гаплотипа на дереве 67-маркерных гаплотипов. Но возможны следущие нисходящие субклады, которые пока не определяли:

Ниже приведено дерево 67-маркерных гаплотипов гаплогруппы I-M170 (гаплотипы взяты из Проекта FTDNA). Всю левую часть дерева занимают гаплотипы гаплогруппы I1-M253, c общим предком 3690±370 лет назад (хотя эта гаплогруппа возникла примерно 27500 лет назад, но она прошла бутылочное горлышко популяции примерно 3500-4000 лет назад, как и все субклады гаплогруппы I в этот период времени, в целом между 4000 и 5000 лет назад), они имеют вид плотной, компактной ветви. Гаплогруппа I2 занимает правую часть дерева, там расположены гаплотипы субкладов I2a, I2b, и I2c, последние – в правой верхней части дерева, и отделены от остальных высокой узкой ветвью, которя к I2c не относится. На представленный гаплотип ID00005 указывает угол квадратика с буквой Х. Эта ветвь, одна из трех ветвей субклада I2c-L596, приведена ниже в увеличенном виде, и представленный гаплотип опять помечен крестиком.
Теперь о ветви из 9 гаплотипов (три из Грузии [под номерами 427, 451 и 452], два из Белоруссии [454 и 455], по одному из Литвы [456], Австрии [453] и Турции [444]). Базовый гаплотип этой ветви (мутационные отличия от представленного гаплотипа отмечены)
14 24 15 10 12 13 11 13 11 13 11 29 – 18 8 8 11 11 24 15 20 32 11 14 15 16 – 11 10 19 21 15 13 17 19 34 36 12 10 12 8 16 16 8 12 10 8 10 9 12 21 21 16 11 12 13 14 8 13 25 20 12 13 11 13 11 12 12 11
Все 9 гаплотипов ветви имеют 123 мутации от этого базового гаплотипа, что означает, что общий предок ветви жил 123/9/0.12 = 114 → 129 условных поколений назад, или 3225±430 лет назад. Представленный гаплотип отличается от базового на 17 мутаций (ряд мутаций считаются по особым правилам), и это дает примерно то же самое время от общего предка, в пределах погрешности расчета. Это показывает, что ID00005 – прямой потомок общего предка ветви гаплогруппы I2c, который жил примерно в конце II тыс. до н.э.

Ниже показаны три основные ветви субклада I2c-L596, которые соответствуют семи его нижестоящим субкладам (см. диаграмму выше). Представленный гаплотип определенно входит в один из них, но в какой – мы пока не знаем. У каждого – своя история, которая пока нам тоже неизвестна (та, что известна, приведена ниже). Как мы видим, география ветви ID00005 весьма обширна – от Австрии, Беларуси и Литвы до Турции и Грузии, но более тонкое подразделение ветвей по нижестоящим субкладам в итоге позволит ее уточнить. Проекты FTDNA в этом отношении пока не помощники, в них этих более тонких субкладов нет, но московская Лаборатория ДНК-генеалогии будет их определять.
Чтобы понять, насколько сложной и трагической была история субклада L596 и его нижестоящих субкладов, заметим, что сам субклад L596 возник примерно 21300 лет назад (как показывает лесенка его снипов в Y-хромосоме), но он в ходе истории Европы то почти полностью погибал, то снова возрождался из немногих выживших носителей L596. В итоге предок подветви ID00005 жил примерно 3225 лет назад, соседней подветви (из семи гаплотипов) на диаграмме выше – примерно 2475 лет назад, верхней подветви (из девяти гаплотипов) – 2800 лет назад, и всех трех ветвей вместе – 5950±645 лет назад. А геномный анализ показывает, что нижестоящий субклад I2c1 был образован 15500 лет назад; следующий ниже субклад, I2c1a-L1251 – 10100 лет назад, про остальные данных пока нет. Мы видим, что пока обнаружены три ветви L596, возраст каждой – около 3200-2500 лет назад.

Как уже говорилось, показанная ветвь по географии смешанная, но с уклоном в Восточную Европу и Кавказ (Австрия, Беларусь, Литва, Грузия, Турция). Вторая ветвь – западная (Англия, Франция, Италия, Нидерланды). Третья – тоже западная (Англия, Шотландия, Ирландия, Дания, Германия).
Итак, подводим итоги по состоянию наших знаний на сегодняшний день. Гаплогруппа I – очень древняя, образовалась более 40 тысяч лет назад, где – пока неизвестно, но скорее всего в Европе. Около 30 тысяч лет назад она разошлась на субклады I1 и I2, и последняя разошлась на субклады I2a, I2b и I2c-L596 примерно 21300 лет назад. Гаплогруппы I и I2 обнаружены в древних ископаемых костных останках в Центральной Европе и в Швеции с датировками 7 тысяч лет назад. Немногим позже жил общий предок трех ветвей I2c-L596, потомки которого живут в настоящее время от Британских островов до Турции. Но примерно 4500 лет назад жизнь носителей гаплогруппы I, как и всех остальных в Европе резко нарушается, и почти все гаплогруппы (кроме R1b) из Европы исчезают, или откатываются на периферийные районы Европы, часть уходит в Переднюю Азию и на Кавказ.
Эти гаплогруппы в виде отдельных представителей проходят долгий и болезненный период выживания, и выжившие начинают приумножаться, формируя общих предков новых ветвей. В их числе были и носители субклада I2c-L596, пошедшие «в рост» 3200-2500 лет назад, то есть в конце II – середине I тыс. до н.э. Почти тогда же стала возрождаться гаплогруппа I2a, общий предок которой жил 2300 лет назад в Восточной Европе, и около 5000 лет назад на Британских островах. Немногим раньше стала возрождаться гаплогруппа I1, с выжившим общим предком примерно 3700 лет назад. В этом отношении субклад I2c-L596 имеет наиболее древние корни в отношении сегодняшних носителей этой гаплогруппы – около 6000 лет назад.
Отрадно, что нижестоящие субклады L596 разделяются на соответствующие ветви гаплотипов (см. диаграмму выше). Это значит, что при выявлении этих нижестоящих субкладов в московской Лаборатории ДНК-генеалогии у достаточного количества человек (например, у полусотни) мы сможем провести более детальное отнесение гаплотипов к субкладам, и более детально разобраться с преимущественной географией субкладов.
Безусловно, будет полезно расширить круг персональных интерпретаций на женские гаплогруппы, и для их тестирования можно анализировать мДНК как у женщин, так и мужчин. Дело в том, что мужчина получает мтДНК с кровью от матери, но дальше мтДНК уже не передает, потому что митохондрии сперматозоидов после оплодотворения (или в ходе него) разрушаются. Сперматозоид содержит около десятка митохондрий, поскольку на вращение хвостиком нужна энергия, а ее поставляют именно митохондрии. Когда сперматозоид достиг цели, то митохондрии отбрасываются. Узнаете мужчин?
В очень редких случаях митохондрия все-таки проскакивает, но это по отношению к организму будущей матери чужая митохондрия, и добром это, как правило, не кончается. А кончается нередко острыми патологиями.
Короче, мтДНК передается от матери дочерям по всей цепочке поколений, десятками, сотнями тысяч лет, миллионами лет, и так далее. А сыновьям – только на одно поколение. Сыновья передают своим сыновьям Y-хромосому, не передают никому мтДНК – ни сыновьям, ни дочерям, но передают всем свои остальные 22 хромосомы, рекомбинантные. Поэтому, повторяю, тесты на мтДНК можно проводить по ДНК как женщин, так и мужчин. В обоих случаях результат один – выявление мтДНК матери и всей предыдущей цепочки мтДНК со всеми накопленными мутациями. То, что это дает – описано во множестве изданий, и, в частности, в наиболее адаптированном для нас виде – в книгах «Интернет. Заметки научного сотрудника» (2010), на стр. 368-379, и в несколько дополненном виде – в книге «Происхождение славян» (2013), стр. 265-284.
Прежде чем начать акцию по массовой персональной интерпретации мтДНК, предлагаем пробный вариант, выполненный одним из коллег. Поскольку вариант тестовый, имя автора интерпретации пока скрыто. На данном этапе важно, чтобы продукт был признан как минимум приемлемым, а лучше – хорошим или даже отличным. Так что это – на рассмотрение потенциальных заказчиков, а также всех читателей.
Итак, в качестве пробного варианта рассмотрим следующий:
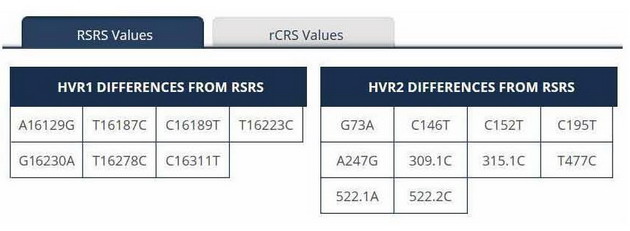
Представленные данные позволяют проследить историю рода на глубине от палеолита (каменного века) лишь до горизонта нескольких тысячелетий до н.э. Это обусловлено тем, что, во-первых, женские митохондриальные ДНК имеют значительно меньшую длину высоковариативных участков, и, следовательно, намного менее информативны. А во-вторых, представленные данные не содержат доступную детализацию – например, субклад Н1с, к которому, как будет показано ниже, относится представленная картина мутаций, содержит более 20 нижестоящих субкладов. Поэтому история рода может быть реконструирована лишь с большей степенью приблизительности, по сравнению с интерпретацией данных по Y-хроосоме. Анализ различий HVR1 и HVR2 с RSRS показывает, что представленный гаплотип относится к митохондриальной гаплогруппе H1с. В силу упомянутых выше причин предоставленный образец совпадает с данными многих других мтДНК: в базе данных familytreedna.com присутствуют носители гаплотипов с идентичными RSRS-значениями по HVR1 и HVR2, причем подавляющее их большинство относится к северной и восточной Европе, например, мтДНК для Anela Sarocka (Литва).

История гаплотипа. Женская гаплогруппа H, предположительно, возникла в Западной Азии около 30 тысяч лет назад, прибыла в Европу около 20-25 тысяч лет назад и распространилась на юго-запад континента во франко-кантабрийский регион, что, скорее всего, соответствует культуре мадлен. В период последнего ледникового максимума 20-13 тысяч лет назад большинство палеолитических поселений Северной и Центральной Европы вымерло, в связи с чем представители гаплогруппы Н в большей степени выжили лишь на севере Испании (поэтому в настоящее время данная гаплогруппа с высокой частотой, более 50%, встречается среди басков, которые сложились в данном регионе при ассимиляции синокавказскими по языку пришельцами местного автохтонного населения). Данная (ископаемая) гаплогруппа была выявлена в пещерах La Pasiega и La Chora (мадлен).
На более поздних горизонтах следует отметить раннее присутствие гаплогруппы в Северной Европе и на Русской равнине, начиная с мезо-неолита – в Сертее (Смоленская область), где в слое Сертея VIII был обнаружен носитель гаплотипа H в сочетании с мужским гаплотипом R1a1 (IV тыс. до н.э.). Данная археологическая культура может рассматриваться как дальняя периферия культуры воронковидных кубков. Также подобные гаплотипы были найдены в захоронениях V-IV тысячелетий до н.э. в донецком регионе и в могильнике Южный Олений Остров (где также была выделена и мужская гаплогруппа R1a1*).
Субклад H1c. Показатель T477C позволяет определить принадлежность гаплотипа к субкладу H1c. Предполагается, что распространение подклассов H1, H3, а также сестринской гаплогруппы V связано с внутриевропейской экспансией во франко-кантабрийском регионе после последнего ледникового максимума около 13 тысяч лет назад. Гаплогруппа H1 составляет значительную долю западноевропейских митохондриальных ДНК, также в большой степени свойственна баскам Испании (28%), португальцам (26%), жителям Андалусии (24%) и этнографической группе пасьего в Кантабрии (24%). В Северной Африке необычно высокая доля гаплогруппы H1 среди туарегов Ливии (61%), что также указывает на смежный с Испанией регион. Также мтДНК гаплогруппа H1 распространена среди других жителей Иберийского полуострова, Северной Африки и Сардинии. При этом ее носители составляют свыше 10% популяции населения во Франции, на Британских островах, в Альпах, во многих регионах Восточной Европе, и не менее 5% в прочих местах Европы.
Возраст гаплогруппы H1c около 9400 лет. И в связи с этим встает вопрос о времени и дате его перемещения в Северную и Восточную Европу. Данный субклад обнаружен у земледельцев культуры воронковидных кубков Скандинавии, 3500-2500 до н.э. (Gokhem2). В предшествующих культурах Скандинавии и Севера Европы подобного гаплотипа не находили. Учитывая появление этого субклада на севере и востоке Европы уже в мезо- неолите, в широкой полосе от Оленьего Острова и до юга Украины позволяет ставить вопрос о миграции с юга. Поскольку франко-испанский регион может рассматриваться как один из центров распространения данной гаплогруппы в предшествующую эпоху, то в качестве логичной гипотезы о появлении этого гаплотипа можно считать версию о проникновении в Северную и Восточную Европу морским путем со Средиземноморского бассейна носителей культуры кардиальной керамики (гипотеза Д.Л. Гаскевича о северопонтийском импрессо).
Она позволяет связать появление носителей H1 за пределами Западной Европы с миграциями (либо иными культурно-археологическими связями) комплекса кардиальной керамики, чьи представители могли передвигаться вдоль побережья на кожаных лодках. Гаскевич даже привел создателей мегалитических культур и кардиальной керамики импрессо Западной Европы и Средиземноморья, в среде которых встречается данный субклад, через существовавший до VI тысячелетия до н.э. Босфорский перешеек в Крым и на юг Украины через буго-днестровскую культуру.
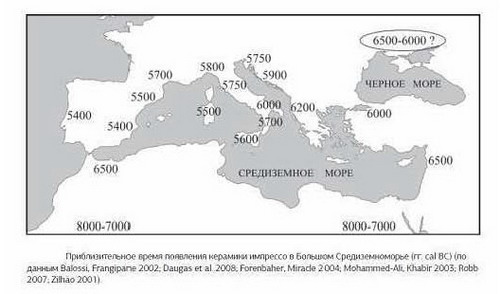
В качестве итога анализа можно констатировать, что представленный гаплотип соответствует найденному в Gokhem2 субкладу представительницы культуры воронковидных кубков, которую многие исследователи считают индоевропейской. Материнская гаплогруппа H была найдена в районе древнего присутствия мужских гаплотипов R1a1 (Южный Олений Остров, Сертея), что позволяет считать эту гаплогруппу исконной для Русской равнины и своеобразным «спутником» мужской R1a1. Более давние связи уводят корни H1c в Средиземноморье.
Для детального исследования родословной заказчика рекомендуется сделать более детальное типирование, поскольку, напоминаем, гаплогруппа H1c содержит свыше 20 различных субкладов. Такое типирование будет вскоре проводиться московской Лабораторией ДНК-генеалогии.
Часть 1: Вводная часть и гаплогруппа R1a
Анатолий А. Клёсов,
доктор химических наук, профессор
Перейти к авторской колонке
 Все выпуски международного научного журнала «Исторический формат» можно бесплатно скачать на официальном сайте. Сейчас готовится к изданию третий номер. Это свободный и открытый проект в стиле Open Science, который имеет научные и просветительские цели. Примите прямое и деятельное участие в выпуске журнала «Исторический формат», поддержите первое в России независимое научное издание по истории, войдите в состав Общественного попечительского совета журнала! Подробнее… Все выпуски международного научного журнала «Исторический формат» можно бесплатно скачать на официальном сайте. Сейчас готовится к изданию третий номер. Это свободный и открытый проект в стиле Open Science, который имеет научные и просветительские цели. Примите прямое и деятельное участие в выпуске журнала «Исторический формат», поддержите первое в России независимое научное издание по истории, войдите в состав Общественного попечительского совета журнала! Подробнее… |


















