Как говаривал вождь, неважно, как голосуют, важно, кто считает. Тот же принцип применим к популяционной генетике – неважно, что там в геноме, важно, кто считает. А считают попгенетики, мы уже знаем, как… Поэтому так важна и интересна каждая статья по геномным исследованиям, особенно если в ней есть гаплогруппы-гаплотипы, потому что мы можем сразу же получить ответы на вопросы, задаваемые авторами в статье (и на наши вопросы), с помощью методов ДНК-генеалогии, поначалу не обращая внимания на геном. И затем объективно сравнить с тем, что получают попгенетики из геномных данных, в тысячи раз более информативных, если их обрабатывать правильно, а не как попгенетики. И тут же становится ясно, что на самом деле более информативно, и кто получает какой ответ на поставленные вопросы.

Поскольку такое объективное сравнение уже много раз проводилось на Переформате, то вот – сухой остаток. Первое – попгенетики не умеют анализировать данные. Имея в руках колоссальный объем информации, предоставляемый геномом, попгенетики умудряются из горы сделать то ли мышь, то ли блоху, причем эта блохомышь оказывается какой-то дефективной, уродливой. На поставленные в начале своей же статьи вопросы ответов не получают, или получают в таком общем, вязком и уклончивом виде, что лучше бы не получали. Или вопросы не задавали. При этом попгенетики заваливают читателя горами букв и цифр, цветных картинок, какими-то «показателями вариабельности», «примесей» (admixtures), «компонентного анализа», и чего у них только нет, но это все квазинаука, потому что сродни тому театральному «что говорить, когда нечего говорить», повторяемому скороговоркой в толпе. Ответов-то они на конкретные вопросы все равно не получают, приводимые в статьях гаплотипы все равно не анализируют, а если какие-то расчеты к ним применяют, то без пояснений, и получая неверные показатели. А ДНК-генеалогия по их же данным, но своими методами, получает четкий и информативный ответ.
 Поддержите проекты Академии ДНК-генеалогии: ваше пожертвование – это дальнейшее изучение истории наших предков, выпуск тематических книг, организация научных мероприятий, исследование палео-днк и ещё многое другое. У нас пока нет других помощников, кроме вас. Поэтому если вы считаете нашу работу полезной, нужной и можете её поддержать, то будем благодарны. Сделать пожертвование от 100 до 5000 руб. можно буквально в один клик по этой ссылке. Поддержите проекты Академии ДНК-генеалогии: ваше пожертвование – это дальнейшее изучение истории наших предков, выпуск тематических книг, организация научных мероприятий, исследование палео-днк и ещё многое другое. У нас пока нет других помощников, кроме вас. Поэтому если вы считаете нашу работу полезной, нужной и можете её поддержать, то будем благодарны. Сделать пожертвование от 100 до 5000 руб. можно буквально в один клик по этой ссылке. |
Поэтому такой сравнительный анализ их статей, с одной стороны, и подходов ДНК-генеалогии, с другой, оказывается поучительным и образовательным. Давайте сейчас этим опять займемся. Опять узнаем новое.
Перед нами – новая статья под названием «Геномные подписи мужской миграции, сформировавшей генетический пул Индии», в журнале Journal of Human Genetics, в авторах – 11 человек, плюс весь Генографический консорциум, около полусотни человек, начиная с Балановских, матери и дитя. Наши пострелы везде поспели, так сказать. Первый автор – GaneshPrasad ArunKumar. В авторах также Spencer Wells, Bennett Greenspan, и прочие не то, чтобы ученые, но люди известные. Однако в попгенетике есть правило, закон такой – чем больше авторов статьи, тем статья более безликая и серая. В общем, это понятно – нет ответственного, по сравнению с тем, когда автор один, или хотя бы двое. Ответственность размазывается, диссипирует. Никто не будет выкладываться, когда бенефициантов много.
Так получилось и на этот раз. Абстракт статьи уже показывает, что «коллективу все равно». Абстракт – это лицо статьи, в нем конкретно излагают, в чем достижение исследования, чем продвинули науку. Когда авторы серенькие, неумелые или безразличные, или попросту неквалифицированные, они в Абстракте помещают не то, сколько они собрали грибов, фигурально говоря, а что они собирали грибы. Так сколько грибов собрали? Да мы собирали…
То же самое и в Абстракте описываемой статьи. Из него совершенно непонятно, в чем наука-то продвинулась, что важного нашли? Вместо этого – «на многие вопросы еще не получены ответы», «Индия, как известно, начала заселяться уже давно», «мы изучали генетическую структуру», и последняя фраза Абстракта – это просто вершина серости – «Мы предлагаем, что древние мужские миграции и оседания (мигрантов) в различных региональных нишах привели к современному сценарию заселения Индии». Ась? Кто-то что другое ожидал? Другой сценарий? Это что, для этого геном надо было изучать?
Продолжим разбор полетов. Во введении к статье формулируются вопросы, на которые надлежит ответить. Но самой первой идет фраза, что человечество вышло из Африки, причем «по южному пути». В статье это больше не используется, никаких данных к этому не дается. Наоборот, показывается, что по «компонентному анализу» индийцы чрезвычайно удалены от африканцев. Ясно, что статья пишется бездумно, по некоему шаблону. Далее во введении отмечается, что есть разные взгляды на то, как заселялась Индия. Видимо, читателю намекают, что ответ будет здесь получен. Указывается, что генетическая история Индии «сложная». Это, конечно, свежая мысль, оригинальная. Как и та, что надо рассматривать структуру популяции наряду с «культурными элементами». Замечательно.
Далее авторы переходят к тому, что при изучении генетической истории Индии есть «конфликтующие результаты». И какие же авторы собираются прояснить? Начинается перечисление. Например, гаплогруппы F*-M89, H-69 и L-M20 «могут иметь автохтонное происхождение», другие, как I-M304, O2a-M95 и O3a3-M117 «могли придти извне», а с R1a-M17 «вопрос вообще не решен, спорный». Здесь даются ссылки на расчеты с использованием «скоростей Животовского» (включая печально известную статью Андерхилла, Животовского, Балановских и др., 2010), и также на статьи, в которых провозглашается заселение Европы из Индии. На самом деле ни то, ни другое неверно, и удивительно, что этого никто из десятков авторов статьи не знает. Популяционная генетика. Введение заканчивается тем, что мтДНК в Индии довольно компактны, в основном из двух гаплогрупп (M и R), а Y-DNA разнообразны, и коррелируют с языковыми семьями. Это, конечно, не так, хотя авторы это далее многократно декларируют. А не так – даже хотя бы потому, что в статье приводятся примеры носителей R1a из племен, джунглей, тибетско-бирманской и австроазиатской языковых групп, и все они говорят на разных языках.
Далее во введении похвально описали тест Geno 2.0, и понятно, почему – в авторах его создатель Spencer Wells. Пообещали, что это поможет решить поставленные вопросы. Введение заканчивается вопросом-надеждой, что геномный анализ позволит найти корреляцию с гаплогруппами.
Далее, в экспериментальной части сообщается, что изучали геном 66 индийцев, к которым добавили еще 12 образцов из Гуджарата, Западная Индия. Мужчин (Y-ДНК) изучали по 23 снипам, женщин (мтДНК) – по 22 снипам. Геномный анализ проводили, разумееется, по многим тысячам снип-мутаций. Я опускаю детали геномного анализа, какие снипы оставляли и какие выбрасывали, увидим по результатам, что в статье получили. Это более красноречиво.
Переходим к результатам исследования. Чтобы было понятно, чего лишает себя геномный анализ, а именно отсутствие в нем знания и понимания гаплотипов (а по сути и гаплогрупп), и подмену их формализованным машинным рассмотрением сотен тысяч геномных снипов, начнем сразу с ДНК-генеалогии в нашем исполнении. В приложении к статье дан список 17-маркерных гаплотипов в нестандартном формате, суммарная константа скорости мутации для которого равна 0.03 на гаплотип за условное поколение в 25 лет. Если перевести выбранные маркеры в формат последовательности FTDNA, то они имеют вид DYS 393, 390, 19, 391, 426, 388, 439, 389-1, 392, 389-2, 458, 437, 448, GATA H4, 456, 438, 635). Из четырнадцати 17-маркерных гаплотипов гаплогруппы R1a1a (по [неряшливому] описанию авторами, на самом деле это, скорее всего, R1a1a1b2a1-L657) в статье шесть принадлежат браминам
13 26 16 11 12 12 10 13 11 18 16 14 20 13 15 11 24
13 25 16 11 12 12 10 13 11 16 16 14 20 13 15 11 23
13 25 16 11 12 12 10 13 12 18 16 14 20 13 15 11 24
13 25 15 11 12 12 10 14 11 17 16 14 20 13 15 11 23
13 25 15 11 12 12 10 13 11 17 16 14 20 12 15 11 23
13 25 16 11 12 12 10 13 11 17 15 14 20 12 15 11 23
Во всех шести гаплотипах имеются 13 мутаций от базового гаплотипа, что дает 13/6/0.03 = 72 → 78 условных поколений (стрелка – поправка на возвратные мутации), то есть 1950±575 лет от общего предка этих шести браминов. Это – начало нашей эры, плюс-минус несколько веков, что, конечно, относительно недавняя группа браминов. Базовый гаплотип их
13 25 16 11 12 12 10 13 11 17 – 16 14 20 13 15 11 23
является почти таким же, как и базовый гаплотип этнических русских субклада R1a-Z280:
13 25 16 11 12 12 10 13 11 17 – 15 14 20 12 16 11 23
отличаясь только на две мутации (четвертый справа маркер 13 в формате статьи равен 12 в формате FTDNA, как знают специалисты), то есть примерно на 2/0.03 = 67 → 72 условных поколений, или 1800 лет. Выяснять, когда жил общий предок этой группы браминов и общего предка этнических русских гаплогруппы R1a-Z280 не так интересно, поскольку брамины поздние, то есть их выборка была не самой удачной. То, что их предок жил примерно 4175 лет назад, скажет нам мало. Это где-то на середине миграционного пути между Русской равниной и Индией.
А вот рассмотрение всей группы индийских R1a из статьи в количестве 14 человек значительно более интересно. Помимо шести браминов, остальные восемь гаплотипов принадлежат индийцам со всех концов Индии, из дравидских племен и дравидских каст, индоевропейцам, тибето-бирманцам и австроазиатским племенам. Но все гаплотипы их походят на браминские и друг на друга:
14 25 15 10 12 12 10 14 11 17 16 14 20 13 15 11 23
13 25 16 11 12 12 11 14 11 17 16 14 20 13 15 11 23
13 25 16 10 12 12 10 13 11 18 15 14 20 13 15 11 23
13 24 16 10 12 12 10 14 11 18 16 14 21 12 15 11 24
13 25 15 10 12 12 10 14 11 17 16 14 20 13 15 11 23
13 25 15 10 12 12 10 13 11 17 17 14 20 13 15 11 23
13 25 15 10 12 12 10 13 11 17 16 14 20 13 15 11 23
13 26 13 10 12 12 10 14 11 17 15 14 20 12 16 9 23
и суммарно все 14 гаплотипов (6 браминских и 8 небраминских) имеют 48 мутаций от того же базового индийского/браминского, а на самом деле арийского базового гаплотипа, уже приведенного выше. Это дает 48/14/0.03 = 114 → 129 условных поколений, то есть 3225±570 лет до общего предка носителей этих 14 гаплотипов – и браминов, и племен в джунглях, и носителей разных языковых групп Индии, всех тех, предки которых были арии, пришедшие в Индию примерно 3500 лет назад, что не противоречит датировке в 3225±570 лет для общего предка по приведенной небольшой выборке. Поскольку общий предок этнических русских гаплогруппы R1a-Z280 жил примерно 4600 лет назад, то с учетом разницы в две мутации между базовыми гаплотипами (см. выше) общий предок этнических русских и описываемой группы потомков индоариев жил примерно (1800+4600+3225)/2 = 4800 лет назад, то есть это и есть общий предок этнических русских за полторы тысячи лет до прибытия их потомков в Индию в виде уже индоариев.
Итак, мы всего лишь в три касания показали, что (1) выборка браминов в обсуждаемой статье недавняя, (2) как брамины, так и индийцы джунглей и низших каст – потомки индоариев, разошедшихся за три с лишним тысячелетия по разным направлениям индийского субконтинента, и (3) предки индоариев вышли с Русской равнины, или прошли по ней, оставив предков этнических русских гаплогруппы R1a или их ближайших родственников примерно 4600 лет назад, и примерно через полторы тысячи лет прибыли в Индию.
Это показали методы и методология ДНК-генеалогии. Как видно, геномный анализ для этого нам не понадобился. Можно надеяться, что геномный анализ это, по меньшей мере, подтвердит, или – скорее всего – пройдет намного дальше. Только вот дальше в чем, конкретно? Давайте посмотрим, вернемся к обсуждаемой статье. Теперь нам уже яснее, ориентиры расставлены. Никакие общие ля-ля уже не пройдут.
Впрочем, раскрывая карты, отметим, что ничего подобного геномный анализ в обсуждаемой статье не показал и близко. Ничего из того, что было показано и рассказано выше в рамках ДНК-генеалогии. Ни про ариев с севера, не говоря о Русской равнине или предках этнических русских как предков индоариев, ни что брамины по мужской линии – потомки тех же общих предков, что и носители гаплогруппы R1a из низших каст и из джунглей, как и носители других языковых групп в Индии. Три тысячи лет – большой срок, народная память могла и потерять сведения, но ведь геномный анализ должен был это показать? Он и показал, только попгенетики не заметили. Не обратили внимания, хотя соответствующие геномные данные просто шли им в руки. Школа у попгенетиков такая, точнее, ее отсутствие. Потеряна способность анализировать данные, или этой способности никогда и не было. Ни Спенсер Уэллс, ни Беннет Гринспэн, ни Балановские, простигосподи, никто из них не умеют видеть того, что им показывают геномные данные. А показывают они в принципе много. Хотя без данных ДНК-генеалогии они во многом бессильны, и этого никак не могут усвоить попгенетики. Что давно надо было просить ДНК-генеалогов – помогите, пожалуйста, мы без вас не справляемся. Это же надо усвоить и историкам, и археологам, и лингвистам, и специалистам по этногенезу.
Для большей полноты картины и сопоставления с данными геномного анализа надо упомянуть, что кроме упомянутых шести браминов гаплогруппы R1a были протестированы еще шестеро, которые оказались гаплогрупп С5, J2a* и L1 (по двое на каждую). Остальные протестированные индийцы, числом 46 человек (в дополнение к описанным выше восьми носителям R1a), распределились по гаплогруппам/субкладам следующим образом, в правой колонке даны оценки времен жизни их общих предков, лет назад:
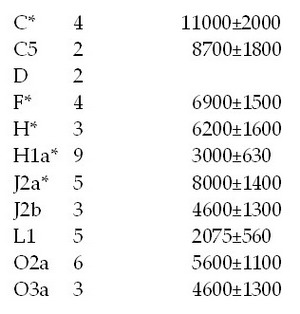
И здесь гаплотипы браминов гаплогрупп С5, J2a* и L1 ничем не отличались от гаплотипов прочих индийцев тех же гаплогрупп. Как видно, почти все перечисленные выше популяции намного старше индоариев, и только две – H1a* и L1 – заметно моложе. Новизны здесь никакой тоже нет, и дерево гаплотипов ниже показывает намного более детальные данные, построенные по 187 гаплотипам Индийского проекта FTDNA. Видно, что ветвь гаплогруппы R1a на дереве слева значительно моложе всех остальных (потому что более компактная и неглубокая). В обсуждаемой статье про индийский геном приведенные выше гаплотипы и датировки тоже не рассматриваются и не обсуждаются. Да и как они могли бы? Авторы статьи не владеют аппаратом и подходами ДНК-генеалогии.
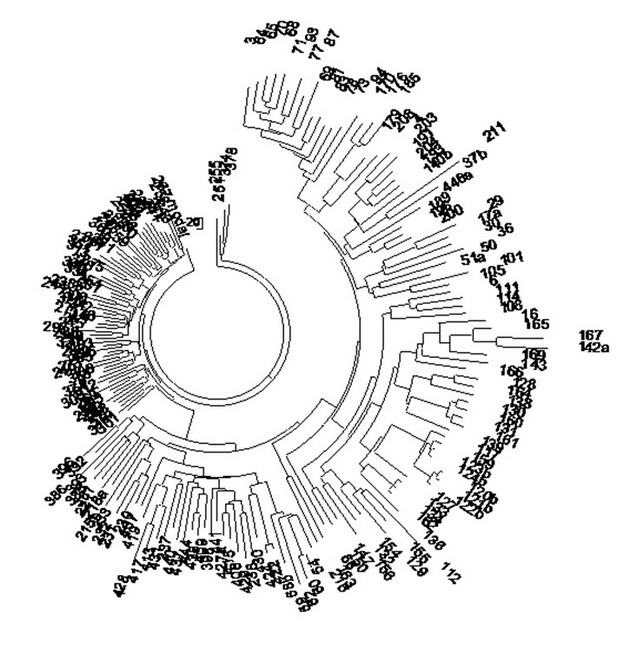
Дерево 187 индийских гаплотипов Y-хромосомы в 37-маркерном формате. Слева – плотная ветвь из 64 гаплотипов гаплогруппы R1a, с общим предком, жившим 4750±500 лет назад; справа и внизу – серии древних индийских гаплотипов гаплогрупп (по часовой стрелке) Н, L, Q, L, J-M304, O, C, L, C, J1-M267, J2b-L282, J2-M172, R2-M124, Q (повторы гаплогрупп – это отдельные ветви этих гаплогрупп). Общие предки ветвей справа и внизу жили 8-12 тысяч лет назад. Построено по данным Индийского проекта FTDNA, Клёсов и др., ДНК-генеалогия как молекулярная история. Математическая морфология. Математический и медико-биологический журнал. Т. 14. Вып. 1. 2015, ссылка.
Что же показал геномный анализ в обсуждаемой статье, и какие выводы были сделаны? Как особенно отмечают авторы статьи, геномный анализ выявил три основных компонента генетического пула Индии – азиатский юго-западный (темно-красный на диаграмме ниже), азиатский юго-восточный (синий), и азиатский северо-восточный (коричневый).
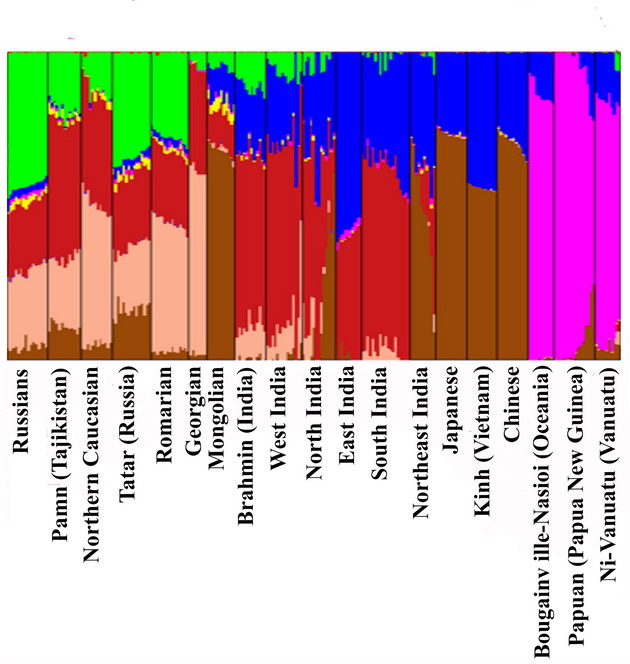
Я не мог найти в статье расшифровку зеленого цвета, которая занимает около половины диаграммы этнических русских, и которой почти нет в Индии, но ее поможет объяснить то, что тот же цвет занимает примерно 20% в Сардинии, у греков, болгар и итальянцев, и примерно 40% у англичан, немцев, испанцев с португальцами, датчан и финнов. Как и у русских и у татар.
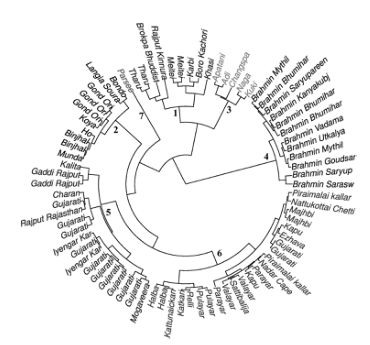
Далее авторы приводят в статье диаграмму, которая, по их сообщению, показывает корреляцию генома с языками, на которых разговаривают носители данного генома. Признаться, я так и не понял, по какому принципу популяции расположились по кругу (в статье это называется «иерархическая кластеризация, основанная на пропорциях» со ссылкой на диаграмму, приведенную выше, и что в качестве критерия применялись территориальная и языковая принадлежность), но пусть это остается за авторами. Занятно, что браминов выделили в отдельный кластер, независимо от того, в каком регионе Индии они проживают, потому что у них «уникальное происхождение». Как мы только что видели, никакого «уникального происхождения» у браминов в той выборке нет. Ну да ладно, думаю, что читатели уже начали кое-что понимать.
Нумерация на диаграмме ниже следующая: (1) говорящие на тибето-бирманских языках из северо-восточной Индии, (2) говорящие на австроазиатских языках из восточной Индии, (3) говорящие на тибето-бирманских языках из северо-восточной и северной Индии (мое примечание – здесь что-то не так, тибето-бирманцы из северо-восточной Индии уже были в п. 1, ну да ладно, нам не привыкать), (4) брамины, говорящие на индо-европейских (ИЕ) языках по всей Индии, (5) не-брамины, говорящие на ИЕ языках по всей Индии, (6) дравиды из южной Индии, (7) популяция парси из западной Индии.
К этому авторы добавили «компонентный анализ», и заверили, что он подтверждает то самое деление на популяции, которые «формируют гетический пул Индии». Ниже – диаграмма того самого «компонентного анализа», на котором стоит вся популяционная генетика:
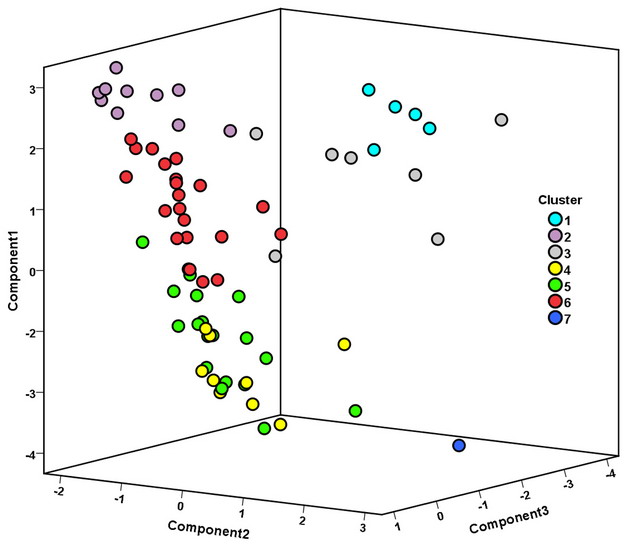
В общем-то, наверное, не удивительно, что геном индийских популяций в разных концах огромной Индии ближе друг к другу в одной части Индии, чем почему-то окажется ближе друг к другу в разных частях Индии, но при чем здесь вопросы, поставленные авторами во введении к статье? Где обещанное (хотя и не всегда в явном виде) прояснение, кто там автохтоны, а кто пришли извне? Где решение вопроса о носителях R1a, который «спорный»? Откуда и когда пришли в Индию арии? Или, напротив, когда они ушли из Индии и в итоге заселили Европу, как утверждают многие индийские авторы, вкупе с примкнувшим к ним Животовским, Кивисилдом и эстонской школой, а также неким «индологом Семененко», в свое время бежавшим с Переформата? Понятно, что «геном большой, ему видней», описывать можно много, но хотелось бы видеть направленный ответ на поставленные вопросы, не так ли?
И вообще, тема браминов осталась нераскрытой. Это – номер 4 на диаграмме выше, желтые кружочки. Да, сидят на диаграмме компонентного анализа плотно, но как сказал бы Станиславский – «не верю!». Если бы там были только брамины гаплогруппы R1a, тогда понятно, их общий предок в выборке статьи жил всего 2000 лет назад (округленно), но их всего шесть человек, а у них в компании еще шесть браминов гаплогрупп С5, J2a* и L1, а они, пардон, уж очень далеки по происхождению от R1a. Как они вообще могут оказаться в одной плотной группе друг с другом, а также в компании с зелеными кружочками не-браминов, говорящих на ИЕ языках по всей Индии, а там, по данным статьи, носители гаплогрупп С*, D, H*, H1a*, J2a*, J2b, L1… Как говорил хрестоматийный герой, «меня начинают терзать смутные подозрения»… И даже приводимые авторами как показатель тесной ассоциации браминов «P-value of 2.95 x 10-33» (??) как-то не убеждают, как и соседние величины P-values 7 x 10-24 и 1.57 x 10-17.
Начинают проясняться методологические особенности исследования, точнее, интерпретаций. Авторы пишут, что дравиды южной Индии показывают максимальную пропорцию азиатского юго-западного компонента (58%), и это, по их мнению, свидетельствует об их автохтонности – или тамошнего происхождения, или экспансии оттуда, то есть из южной Индии. Вполне возможно. Только вот проблема – геном не укоренен, и такие выводы делать нельзя. Если непонятно – то как быть с браминами? В том числе гаплогруппы R1a? У них – практически такая же доля (55%) того же самого азиатского юго-западного компонента (см. цветную диаграмму выше, а число дается в статье), они что, тоже автохтоны южной Индии? Да и вообще счет таких показателей с точностью до одного процента без указания погрешностей опять заставляет вспомнить об отсутствии у попгенетиков научной школы.
Вы будете смеяться, но мы уже приближаемся к завершению раздела «Результаты». Авторы продолжают рассуждения на уровне «южные и западные индийские популяции более близки (видимо, по геному) к популяциям, говорящим на ИЕ языках (брамины, или Италия), а северные и северо-восточные индийские популяции показывают большую близость к азиатским северо-восточным популяциям (Китай)». Это все, конечно, чрезвычайно научно, но где ответы на прямые, исходные вопросы? Как замечаете, статья вовсе не о том. Она имеет типичный попгенетический стиль – что-то описывать, описывать, и далее еще описывать, но не отвечать на поставленные вопросы.
Последняя часть раздела «Результаты» посвящена обсуждению – наконец – снипов. И действительно, зачем тогда они их определяли? Но, оказывается, определяли опять же для построения диаграмм «компонентного анализа». Пишут, что снипы определяли с помощью Geno 2.0, но в статье их не показали, кроме самых поверхностных, типа R1a1. А вот и то, зачем определяли снипы – диаграмма компонентного анализа:
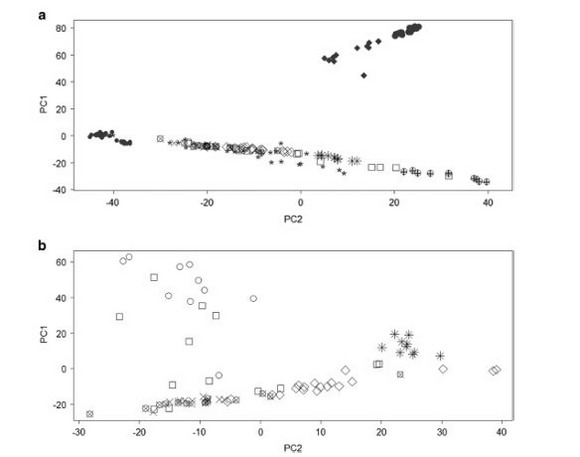
Кто что понял? Хорошо, вот объяснения: наверху – «глобальный компонентный анализ», ниже – «индийский компонентный анализ». Символы отражают географию образцов. Крестики – брамины, квадратики – северная Индия, ромбики – южная Индия, звездочки – восточная Индия, кружочки – северо-восточная Индия, крестики в кружочках – западная Индия, маленькие закрашенные кружочки – европейцы, закрашенные ромбики – афроамериканцы, закрашенные кружки – африканцы, закрашенные звездочки – южная Америка, плюсики – восточная Азия.
Поскольку смысл это все имеет небольшой, послушаем выводы авторов. Итак, верхняя диаграмма показывает, что индийцы оказываются между европейцами и азиатами. Замечательно. Это, конечно, науку продвигает. А что еще авторы увидели? То, что африканские символы (то есть снипы) находятся в стороне. Это – черные символы в самом верху. Все впечатлены. Видимо, действительно стоило делать снипы на Geno 2.0. А что еще? Авторы дают еще три вывода, ради чего все это, видимо, делалось: (1) выявлено отделение индийских образцов от европейских, азиатских, африканских и американских индейцев, (2) индийские образцы ближе к азиатским и европейским, чем к африканцам и американским индейцам, и (3) наличие кластеров внутри индийских образцов. Опять, никакого отношения к поставленным ранее прямым вопросам это не имеет.
Раздел «Результаты» заканчивается двумя положениями, одно из которых меня порадовало. А именно, авторы заключили, что есть корреляция между гаплогруппами Y-хромосомы и всем геномом. Я об этом пишу последние как минимум пять лет, в том числе и на Переформате, наиболее обстоятельно, видимо, в статье Действительно ли «генетики нашли разных русских»? в марте 2013 года. Моя статья на Переформате заканчивалась так – это «позволяет понять, что лежит в основе вариаций в геноме, задающих глубинные различия между популяциями. Эти различия идут из тьмы тысячелетий, и что особенно интригует – они определенно завязаны на мутации, определяющие мужские гаплогруппы. Это – совершенно новая концепция, и решать эту загадку надо в содружестве генетиков и ДНК-генеалогов».
Правда, авторы обсуждаемой статьи про геном это свое наблюдение не развили, ограничившись небольшой фразой. А если честно, то и не показали, это просто их соображение, или догадка. Но уже хорошо.
Второе соображение тоже справедливо, об этом я также многократно писал, а именно, что мтДНК в изученной работе оказались неинформативными. Авторы приписывают это «патриархальной социальной практике», что, скорее всего, так и есть.
Всё, раздел «Результаты» закончен. Раздел «Дискуссия», как водится у погенетиков, вязкий и неинтересный. Ничего нового уже нет. Под «дискуссией» у попгенетиков понимают повторение раздела «Результаты», только более длинно. Там есть вкрапления, вообще не относящиеся к вопросам, поставленным в начале статье, хотя, как мы уже знаем, на них ответов не только не было, но и отвечать не пытались. «Что говорить, когда нечего говорить». Авторы, например, пишут, что «Носители ИЕ языков в северной Индии имеют различное предковое происхождение. Шесть из 11 носителей ИЕ с севера Индии живут вблизи от говорящих на тибето-бирманских языках в гималайском регионе. Таким образом, у них есть небольшая генетическая близость, которая оттягивает индо-европейцев севера (Индии) от других индоевропейцев (Индии)». Что это? К чему это? Как это отвечает на поставленные в статье вопросы? Ведь в таком стиле можно продолжать до бесконечности, не отвечая ни на единый вопрос.
В качестве заключения авторы описывают следующие «наблюдения», которые считают, видимо, наиболее важными в своей работе, и представляя их как «сценарии»: (1) индийский генетический пул состоит из «нативных» (видимо, имеются в виду автохтонных) индийцев, (2) вариации в геноме коррелируют как с географической, так и с языковой вариациями, (3) корреляция гаплогрупп Y-хромосомы с геномными вариациями (которая так и не была продемонстрирована, это скорее рассуждение – ААК) позволяет предположить, что наибольшее влияние на формирование генетического пула Индии оказали мужские миграции, и (4) отсутствие корреляции мтДНК и генома позволяет предположить, что женская генетика в Индии преодолевала географические и лингвистические барьеры. И последняя фраза заключения, видимо, самая важная: «Мы предполагаем, что древние мужские миграции и заселение различных региональных ниш привело к современному сценарию и заселению Индии». Фраза косноязычна даже по-английски, и тем более в переводе. Но что есть, то есть. Что говорить, когда нечего говорить.
Анатолий А. Клёсов,
доктор химических наук, профессор
Перейти к авторской колонке
 Поддержите проекты Академии ДНК-генеалогии: ваше пожертвование – это дальнейшее изучение истории наших предков, выпуск тематических книг, организация научных мероприятий, исследование палео-днк и ещё многое другое. У нас пока нет других помощников, кроме вас. Поэтому если вы считаете нашу работу полезной, нужной и можете её поддержать, то будем благодарны. Сделать пожертвование от 100 до 5000 руб. можно буквально в один клик по этой ссылке. Поддержите проекты Академии ДНК-генеалогии: ваше пожертвование – это дальнейшее изучение истории наших предков, выпуск тематических книг, организация научных мероприятий, исследование палео-днк и ещё многое другое. У нас пока нет других помощников, кроме вас. Поэтому если вы считаете нашу работу полезной, нужной и можете её поддержать, то будем благодарны. Сделать пожертвование от 100 до 5000 руб. можно буквально в один клик по этой ссылке. |


















